DEPDC5
DEPDC5(またはDEPドメインを含む5 )は、機能が十分に理解されていないヒトタンパク質ですが、いくつかの研究で癌と関連しています。 22番染色体上にある同名の遺伝子によってコードされています。 DEPDC5 識別子
エイリアス
DEPDC5、DEP.5、FFEVF、5を含むDEPドメイン、FFEVF1、5を含むDEPドメイン、GATOR1サブコンプレックスサブユニット
外部ID
OMIM:614191 MGI:2141101 HomoloGene:34718 GeneCards:DEPDC5
遺伝子の位置(ヒト) Chr。 22番染色体(ヒト)
バンド
22q12.2-q12.3
始める
31,753,867 bp
終わり
31,908,033 bp
遺伝子の位置(マウス) Chr。 5番染色体(マウス)
バンド
5 B1 | 5 17.35 cM
始める
32,863,701 bp
終わり
32,994,236 bp
RNA発現パターン Bgee トップ表現
滑膜
大脳皮質
腓腹神経
後腎糸球体 肺 大腿四頭筋
肝臓
前頭極
その他の参照発現データ BioGPS 該当なし
遺伝子オントロジー
分子機能
GO:0032403タンパク質含有複合体結合
GO:00005097、GO:00005099、GO:0005100GTPaseアクチベーター活性
細胞成分
細胞質
細胞質の核周辺領域
GATOR1コンプレックス
サイトゾル
リソソーム
リソソーム膜 膜 Cul3-RINGユビキチンリガーゼ複合体
細胞内解剖学的構造
生物学的プロセス
TORシグナル伝達の負の調節
アミノ酸欠乏に対する細胞応答
GO:0007243細胞内シグナル伝達
GO:0032320、GO:0032321、GO:0032855、GO:0043089、GO:0032854GTPase活性の正の調節
TORC1シグナル伝達の負の調節
オートファジーの調節
出典:Amigo / QuickGO
オーソログ
種族
人間
ねずみEntrez9681 277854 Ensembl ENSG00000100150 ENSMUSG00000037426 UniProt O75140 P61460
RefSeq(mRNA)
NM_001007188 NM_001136029 NM_001242896 NM_001242897 NM_014662
NM_001363852 NM_001363854 NM_001364318 NM_001364319 NM_001364320 NM_001369901 NM_001369902 NM_001369903
NM_001025426 NM_001170567 NM_177786 NM_001360014
RefSeq(タンパク質)
NP_001007189 NP_001129501 NP_001229825 NP_001229826 NP_055477
NP_001350781 NP_001350783 NP_001351247 NP_001351248 NP_001351249 NP_001356830 NP_001356831 NP_001356832
NP_001020597 NP_001164038 NP_808454 NP_001346943
場所(UCSC)
22番染色体:31.75 – 31.91 Mb
Chr 5:32.86 – 32.99 Mb
PubMed検索
ウィキデータ
人間の表示/
マウスの表示/編集
コンテンツ
1 関数
2 遺伝子
3 構造
3.1 ドメイン
3.1.1 DEP
3.1.2 DUF 3608
3.2 二次構造
4 相同性
4.1 オーソログ 4.2 パラログ
5 表現
6 翻訳後修飾
7 交流
8 参考文献
関数
DEPDC5の機能はまだわかっていませんが、 DEPDC5のDEPドメインとDishevelled-1( DVL1 )の間の相同性に基づく細胞内シグナル伝達に関与しているとされています。
この遺伝子の変異は、部分発作の症例に関連しています( doi:10.1038 / ng.2601)。
遺伝子
ホモサピエンスでは、DEPDC5遺伝子は、 PRRL14遺伝子とYWHAH遺伝子の間の22番染色体22q12.2-q12.3の長腕に局在しています。この遺伝子の臨床的関連性には、2倍の肝細胞癌リスクの増加に関連しているイントロンSNP(rs1012068)が含まれます。

構造
ドメイン

DEP
DEPドメインの名前は、 Disheveled、Egl-10、およびPleckstrinのタンパク質に由来し、それぞれにこのドメインのバリアントが含まれています。 82残基にまたがり、 C末端から343アミノ酸です。SWISS-MODELは、ドメイン内に含まれる2つのベータシートと3つのアルファヘリックスを予測します。
その正確な機能は不明ですが、 NCBIでCBLASTを実行すると、DEPDC5DEPドメインはDVL1のDEPドメインと構造的に最も類似しています。アラインメントは1.00e-08のEvalueをスコアリングし、2つのタンパク質のDEPドメイン間の30%の同一性を示します。DVL1では、DEPドメインは、 Wntシグナル伝達経路の一部として原形質膜へのタンパク質の局在化に関与しています。
DUF 3608
DUF 3608ドメインは、 N末端から99アミノ酸の位置にあり、それ自体が280アミノ酸にまたがっています。PELEは、このドメイン内に少なくとも1つのベータシートと2つのアルファヘリックスを予測します。また、26の高度に保存された残基と、いくつかの翻訳後修飾が含まれています。両方の発生については、の後半で説明します。
DUF 3608の機能の証拠は、酵母 ホモログ Iml1pで明らかにされています。Imlp1のDUF3608は、2つのタンパク質パートナーであるNpr2とNpr3への結合を助けると考えられています。一緒に、これらの3つのタンパク質はIml1-Npr2-Npr3複合体を形成し、「非窒素飢餓」オートファジー調節に関与しています。これを発見した研究者は、DUF 3608の名前をRANSに変更することを提案しています(非窒素飢餓条件下で誘発されるオートファジーに必要)。
二次構造
二次構造予測ツールPELEによる全会一致のコンセンサスに基づいて、DEPDC5には少なくとも10個のアルファヘリックスと9個のベータシートが含まれています。これらの二次構造の位置は、下の画像に示されています。赤いハイライトはアルファヘリックスで、青いハイライトはベータシートです。
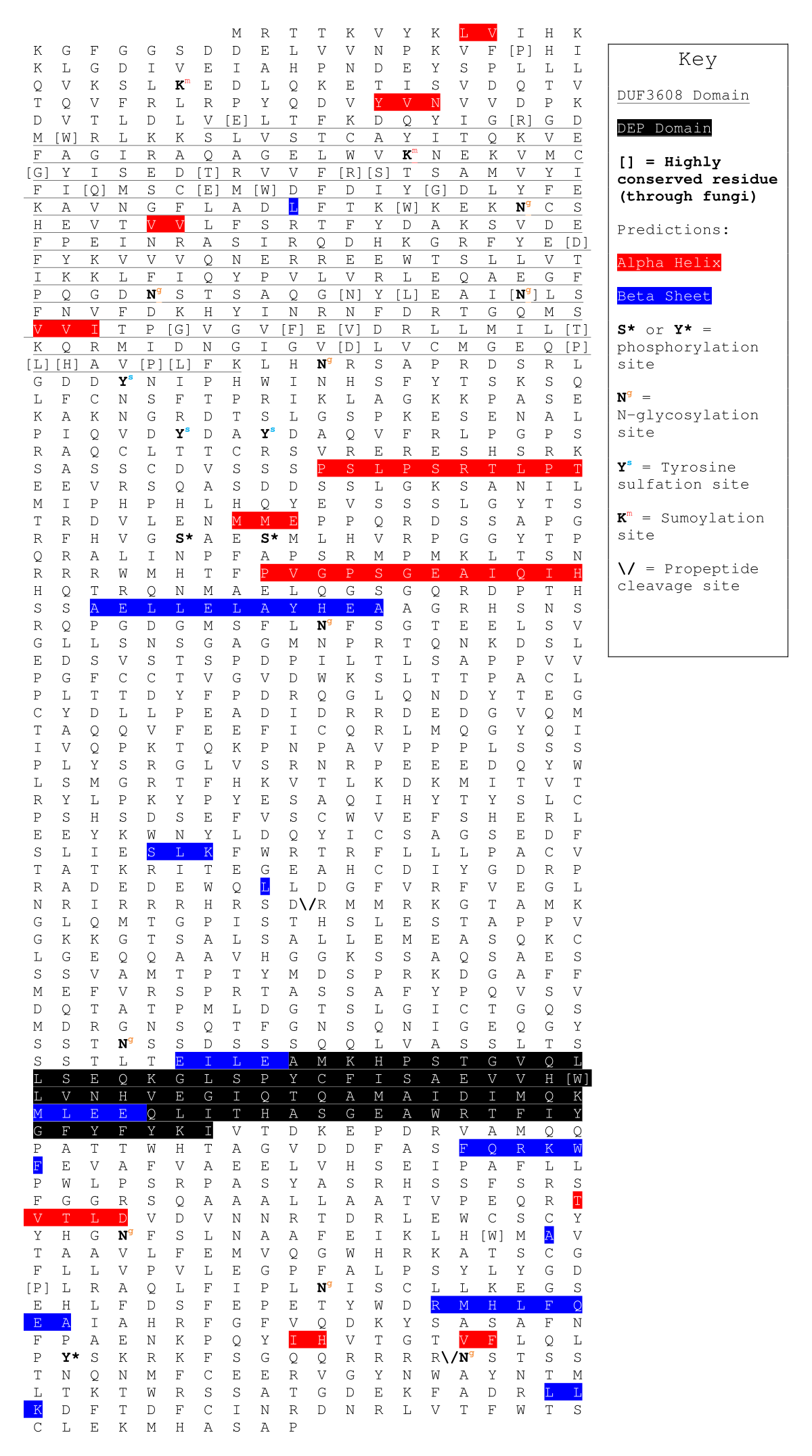
相同性
オーソログ
真菌は、 SaccharomycescerevisiaeやAlbugolaibachiiなど、ヒトDEPDC5とオーソロガスなタンパク質を含む最も遠縁の生物です。真菌では、タンパク質名はIml1p、または液胞膜関連タンパク質Iml1です。他の生物の名前の逸脱には、CG12090(ショウジョウバエ)とAGAP007010(蚊)が含まれます。シクリッドの74%の同一性からチンパンジーの99%の同一性まで、人間と他の脊椎動物種の間で高い保存性が
次の表は、ヒトDEPDC5にオーソロガスな20のタンパク質の分析をまとめたものです。
種族 一般名 NCBIアクセッション番号 NCBI名 長さ シーケンスアイデンティティ 配列の類似性 人間からの発散からの年数(mya)
チンパンジー
チンパンジーXP_003317262 DEPDC5 1572 aa 99% 99% 6.4 Nomascus leucogenys
テナガザルXP_003258163 DEPDC5 1602 aa 99% 99% 20.4
ハツカネズミ
ねずみNP_001164038 DEPDC5 1591 aa 94% 96% 92.4
ボスタウルス牛 XP_002694678 DEPDC5 1593 aa 94% 96% 94.4
ソレックスアラネウス
トガリネズミACE77702 DEPDC5 1570 aa 94% 96% 94.4
モノデルフィスドメスティカ
ポッサムXP_001378772 DEPDC5 1522 aa 89% 93% 163.9
ヤケイ鶏 XP_415249 DEPDC5 1592 aa 88% 93% 301.7 Meleagris gallopavo 七面鳥XP_003211073 DEPDC5 1592 aa 88% 93% 301.7 Taeniopygia guttata
キンカチョウXP_002199825 DEPDC5 1572 aa 87% 92% 301.7
ネッタイツメガエル
カエル XP_002931964 DEPDC5のような1574 aa 79% 86% 371.2
ダニオレリオ
ゼブラフィッシュ XP_691450 DEPDC5のような1590 aa 75% 84% 400.1 Oreochromis niloticus
シクリッドXP_003459226 DEPDC5 1577 aa 74% 82% 400.1 Strongylocentrotus purpuratus
うに XP_794020 DEPDC5に似ています1608 aa 43% 57% 742.9
キイロショウジョウバエ
ショウジョウバエNP_647618 GC12090 1471 aa 41% 57% 782.7
ヒトジラミ
シラミ XP_002429401 DEPDC、推定1538 aa 38% 53% 782.7
ガンビエハマダラカ蚊 XP_308760 AGAP007010-PA 1640 aa 36% 51% 782.7
豚回虫
カイチュウADY40551 DEPDCp5 1359 aa 31% 51% 937.5 Ustilago maydis
トウモロコシ黒穂病 XP_757759 液胞関連タンパク質Iml11867 aa 23% 52% 1215.8 Saccharomyces cerevisiae
酵母NP_012672 Iml1p 1584 aa 20% 50% 1215.8
アルブ ゴライバチイ
白さび CCA27519 液胞膜関連タンパク質推定1591 aa 20% 46% 1362
動物と真菌が分岐して以来、30残基が保存されており、そのうち26残基はDUF3608ドメインに次のマルチプルアラインメントは、 DUFドメインのこの保存を示しています。無脊椎動物および真菌の分岐群の代表は、完全に保存された残基が緑色になっているヒトDUF3608に整列しています。

パラログ
既知のヒトDEPDC5パラログはありませんが、相同なDEPドメインを含む64のヒトタンパク質が酵母タンパク質Iml1、ヒトDEPDC5の最も遠縁のオルソログについても同定されたパラログはありません。
表現
DEPDC5の発現は、RT-PCR分析によってヒト組織に遍在していること、および下のグラフに示すようにDNAマイクロアレイ研究で特徴づけられています。
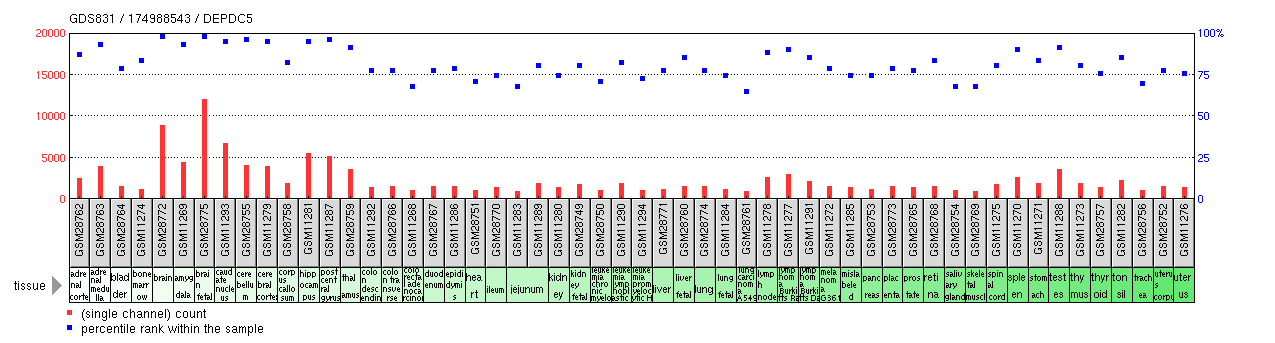
肝細胞癌の患者に関する1つの研究では、非腫瘍組織よりも腫瘍組織で高いDEPDC5発現が見られました。逆に、3つの遺伝子のホモ接合性欠失(1つはDEPDC5)が2つの膠芽腫症例で発見された。他の発現異常には、MDA-MB-231 乳がん 細胞株でのゼロ発現およびP116(ZAP70陰性)細胞株での低発現が含まれます。
翻訳後修飾
以下の翻訳後修飾は、 ExPASy およびPhosphoSitePlus でコンパイルされたヒトDEPDC5タンパク質のプロテオミクスツールを使用して予測されました。
翻訳後修飾 番号/場所 ソース
リン酸化
133 /(Ser:87 Thr:23 Tyr:23) NetPhos 6 / S579、S582、S1499、Y1515、Y1519、Y1543 PhosphoSite Plus 糖化
29 / 5、8、13、14、28、34、56、59、64、93、131、147、229、247、256、319、436、528、609、710、862、878、1008、1185、 1233、1387、1408、1499、1567、1597 NetGlycate N-グリコシル化部位
9 / N201、N298、N311、N384、N684、N1157、N1377、N1444、N1529 NetNGlyc 硫酸化
3 / Y397、Y459、Y462
スルフィネーター SUMO化 2 / K59、K147 SUMOsp プロペプチド切断
2 / R1004-M1005、R1528-N1529 ProP O-グリコシル化0 NetOGlyc
C-マンノシル化0 NetCGlyc
ミリストイル化 0 ミリストイル化
プレニル化0 PrePS
アセチル化 0 NetAcet
交流
DEPDC5は、共免疫沈降および転写因子MYCによって証明されるように、プロテアソームサブユニットPSMA3と相互作用する可能性が DEPDC5は、 NPRL2およびNPRL3との「GATOR1」複合体に含まれています。
参考文献
^ GRCh38:Ensemblリリース89:ENSG00000100150 – Ensembl、2017年5月 ^ GRCm38:Ensemblリリース89:ENSMUSG00000037426 – Ensembl、2017年5月 ^ 「HumanPubMedリファレンス:」。国立バイオテクノロジー情報センター、米国国立医学図書館。
^ 「マウスPubMedリファレンス:」。国立バイオテクノロジー情報センター、米国国立医学図書館。
^ Miki D、Ochi H、Hayes CN、他。。「DEPDC5遺伝子座の変動は、慢性C型肝炎ウイルスキャリアにおける肝細胞癌への進行と関連しています」。ナット Genet。43(8):797–800。土井:10.1038 /ng.876。PMID21725309。_ S2CID205357903。_ ^ Seng TJ、Ichimura K、Liu L、Tingby O、Pearson DM、Collins VP。「マイクロサテライトと22番染色体タイルパスアレイ分析を使用して特定された星状細胞腫における複雑な22番染色体再配列」。遺伝子染色体がん。43(2):181–93。土井:10.1002 /gcc.20181。PMID15770670。_ S2CID45003453。_ ^ “”GeneCards:5を含むDEPドメイン””。
^ “”AceView:ホモサピエンスの複雑な遺伝子座DEPDC5、5を含むDEPドメインをエンコード””。
^ 「SWISS-MODEL」。
^ 「NCBI:CBLAST」。
^ Pan WJ、Pang SZ、Huang T、Guo HY、Wu D、Li L。「dishevelled-1の3つのドメインの機能の特徴づけ:DEPドメインはdishevelled-1の膜移行に関与しています」。CellRes。14(4):324–30。土井:10.1038 /sj.cr.7290232。PMID15353129。_ ^ 「生物学ワークベンチ:PELE」。
^ Wu X、Tu BP。「窒素飢餓がない場合のIml1-Npr2-Npr3複合体によるオートファジーの選択的調節」。モル。Biol。セル。22(21):4124–33。土井:10.1091 /mbc.E11-06-0525。PMC3204073。_ PMID21900499。_ ^ “”NCBI””。
^ 「TimeTree」。
^ 「生物学ワークベンチ:ClustalW」。
^ Civera C、Simon B、Stier G、Sattler M、Macias MJ。「ヒトプレクストリンDEPドメインの構造とダイナミクス:新規DEPドメインサブファミリーの異なる分子的特徴」。タンパク質。58(2):354–66。土井:10.1002 /prot.20320。PMID15573383。_ S2CID45722575。_ ^ 石川K、長瀬T、須山M、他。(1998年6月)。「未確認のヒト遺伝子のコード配列の予測。X。invitroで大きなタンパク質をコードできる脳からの100個の新しいcDNAクローンの完全な配列」。DNA解像度。5(3):169–76。土井:10.1093 / dnares /5.3.169。PMID9734811。_ ^ Johnson JM、Castle J、Garrett-Engele P、他 。「エクソンジャンクションマイクロアレイを用いたヒト代替プレmRNAスプライシングのゲノムワイド調査」。科学。302(5653):2141–4。土井:10.1126 /science.1090100。PMID14684825。_ S2CID10007258。_ ^ Cappellen D、Schlange T、Bauer M、Maurer F、Hynes NE。「新規c-MYC標的遺伝子は、細胞増殖と遊走に対する異なる効果を媒介します」。EMBO担当者。8(1):70–6。土井:10.1038 /sj.embor.7400849。PMC1796762。_ PMID17159920。_ ^ Roose JP、Diehn M、Tomlinson MG、他。。「ErkおよびAblキナーゼを介したT細胞受容体非依存性の基礎シグナル伝達はRAG遺伝子発現を抑制します」。PLOSBiol。1(2):E53。土井:10.1371 /journal.pbio.0000053。PMC261890。_ PMID14624253。_
![]()
^ 「ExPASyプロテオミクスツール:翻訳後修飾予測」。
^ 「PhosphoSitePlus:DEPDC5」。
^ Lim J、Hao T、Shaw C、他。。「プルキンエ細胞変性のヒト遺伝性運動失調および障害のためのタンパク質間相互作用ネットワーク」。セル。125(4):801–14。土井:10.1016 /j.cell.2006.03.032。PMID16713569。_ ^ Zeller KI、Zhao X、Lee CW、他。。「ヒトB細胞におけるc-Myc結合部位と標的遺伝子ネットワークのグローバルマッピング」。Proc。国立 Acad。科学 アメリカ。103(47):17834–9。土井:10.1073 /pnas.0604129103。PMC1635161。_ PMID17093053。_ ^ Bar-Peled、L; Chantranupong、L; チェルニアック、AD; チェン、WW; オッティナ、KA; ブリティッシュコロンビア州グラビナー; 槍、ED; カーター、SL; マイヤーソン、M; サバティーニ、DM(2013)。「mTORCにアミノ酸の十分性を知らせるRagGTPaseのGAP活性を持つ腫瘍抑制複合体」。科学。340(6136):1100–6。土井:10.1126 /science.1232044。PMC3728654。_ PMID23723238。_ “