I-CreI
I- Cre Iはホーミングエンドヌクレアーゼであり、その遺伝子は単細胞緑藻の一種であるクラミドモナスの葉緑体ゲノムで最初に発見されました。それは次の事実にちなんで名付けられました:それはイントロンに存在します。C lamydomonasreinhardtiiから分離されました。これは、 C。reinhardtiiから単離された最初の(I )そのような遺伝子でした。その遺伝子は、 C。reinhardtiiの23SリボソームRNA遺伝子のグループIイントロンに存在します。葉緑体、およびI- Cre Iは、そのmRNAが23S遺伝子の一次転写産物からスプライシングされた場合にのみ発現します。ホモ二量体として機能するI- CreI酵素は、二本鎖DNAの22ヌクレオチド配列を認識し、特定の位置で各鎖の1つのホスホジエステル結合を切断します。I- Cre Iは、ホーミングエンドヌクレアーゼのLAGLIDADGファミリーのメンバーであり、そのすべてが、結合ドメインと活性部位に寄与する保存されたLAGLIDADGアミノ酸モチーフを持っています。I- Cre Iを含むイントロンがイントロンを欠く23S遺伝子に遭遇すると、I- Cre I酵素は23Sの「イントロンマイナス」対立遺伝子に「ホーム」し、その親イントロンをイントロンマイナス対立遺伝子に挿入します。この動作をするイントロンは、モバイルイントロンと呼ばれます。I- Cre Iは、ホストに利益を与えずに独自の伝播を提供するため、利己的DNAの例です。
DNAエンドヌクレアーゼI-CreI
識別子
生命体 Chlamydomonas reinhardtii シンボル ?UniProt 05725 検索する
構造
スイスモデル
ドメイン
InterPro
コンテンツ
1 発見
2 伝播のメカニズム
3 構造研究と可能な用途
4 参考文献
発見
I- Cre Iは、 C。reinhardtii葉緑体ゲノムの23SrRNA遺伝子に介在する配列として最初に観察されました。 23S遺伝子はRNA遺伝子であり、その転写産物がタンパク質に翻訳されないことを意味します。RNAとして、それはリボソームの大きなサブユニットの一部を形成します。この23Sイントロンには、163アミノ酸のタンパク質をコードするオープンリーディングフレームが見つかりました。これは、タンパク質が可動性イントロンのホーミング挙動を促進する可能性があることを示唆しています。さらに、予測されたタンパク質はLAGLIDADGモチーフを持っていました。これは、グループIの可動イントロンでコード化された他のタンパク質に存在する保存されたアミノ酸配列です。1991年の研究により、ORFはDNAエンドヌクレアーゼI- Cre Iをコードし、23S一次転写産物からイントロンがスプライシングされる場所に対応する部位を選択的に切断することが確立されました。この研究はまた、イントロンがまだそれを持っていない23S対立遺伝子に侵入することができたことを示しました。
伝播のメカニズム
I- Cre Iは、I- CreIを含むイントロンを欠く23SリボソームRNA遺伝子の対立遺伝子に存在する22ヌクレオチドのDNA配列を切断するように進化しました。このような「イントロンマイナス」対立遺伝子が切断されると、細胞内で二本鎖切断修復の経路が活性化されます。細胞は、責任のあるI- Cre I酵素を生成した23S対立遺伝子を修復するためのテンプレートとして使用し、 I- CreIを含むイントロンを複製します。結果として生じる「イントロンプラス」対立遺伝子は、I- Cre I酵素の完全なホーミング部位をもはや含まないため、切断されません。このイントロンは、宿主に利益を与えることなく独自の複製を提供するため、I- CreIは利己的DNAの一種です。
構造研究と可能な用途
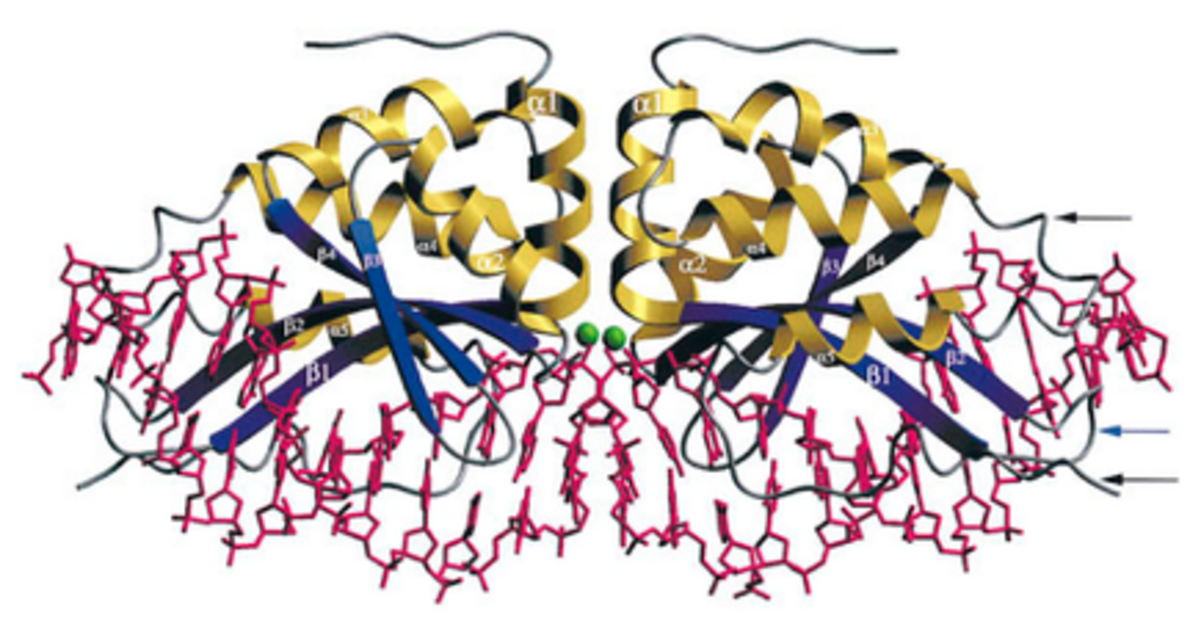
I- CreIのX線結晶構造は
その本来のホーミングサイトに結合しました。
I- Cre Iは、このような長いDNA配列を切断するように進化したため、通常4または6ヌクレオチド配列を切断する制限エンドヌクレアーゼとは異なり、非常に大きなゲノム内の単一部位を切断することができます。4または6ヌクレオチド配列は、偶然に数百万または数十億ヌクレオチドのゲノムで何度も発生すると予想されますが、22ヌクレオチド配列は1回だけ発生する可能性があります(10 9/4 6 vs. 10 9 / 4 22)。I- Cre I切断のこの特異性により、I- CreIは遺伝子ターゲティングの有望なツールになります。ある遺伝子の対立遺伝子の欠陥が原因で病気にかかった場合、その対立遺伝子を機能的な対立遺伝子に置き換えることができれば便利です。I- Cre Iに欠陥のある対立遺伝子のみでDNAを切断させ、同時に細胞が修復テンプレートとして使用する正常な対立遺伝子を提供できる場合、患者自身の相同組換え機構により、機能不全の対立遺伝子の代わりに目的の対立遺伝子を挿入できます。 。I- Cre Iの特異性により、目的の遺伝子の外側での二本鎖切断による有害な影響を減らすこともできます。
このようにI- CreIをツールとして使用するには、本来のホーミングサイトとは異なるDNAの配列を認識して切断する必要がI- CreI構造とそのホーミング部位特異性との関係を研究するための大腸菌遺伝子システムは1997年に作成されました。 1997年にI-CreIタンパク質の構造が決定され、1998年にそのネイティブDNAホーミングサイトにバインドされた結晶構造が解決され、タンパク質のホーミングサイト認識を変更する研究を大いに助けました。その後、ホーミング部位の特異性が変化した変異型のタンパク質が作成されました。 Saccharomyces cerevisiaeの遺伝子システムも作成されており、ホーミング部位の特異性が変更された追加のI- CreI変異株が生成されます。
I- Cre Iは、非常に人気のある真核生物のモデル生物であるキイロショウジョウバエで相同組換えを誘導するためにすでに成功裏に使用されています。分子生物学的手法の進歩と、I- Cre I由来の新規エンドヌクレアーゼのライブラリーの生成により、最終的には病因的に重要な多くの遺伝子の標的化が可能になると思われます。
参考文献
^ Rochaix、JD; Malnoe、P(1978)。「クラミドモナスの葉緑体リボソームDNAの解剖学」。セル。15(2):661–670。土井:10.1016 / 0092-8674(78)90034-x。PMID719757 。_ S2CID31637691 。_ ^ DürrenbergerF 、Rochaix JD(1991年11月)。「Chlamydomonasreinhardtiiの葉緑体リボソームイントロン:invitro自己スプライシング、DNAエンドヌクレアーゼ活性およびinvivo移動性」。EMBOジャーナル。10(11):3495–501。土井:10.1002/j.1460-2075.1991.tb04913.x。PMC453078。_ PMID1915304。_ ^ DürrenbergerF、Thompson AJ、Herrin DL、Rochaix JD(1996年9月)。「クラミドモナス葉緑体における二本鎖切断誘導組換え」。核酸研究。24(17):3323–31。土井:10.1093 / nar/24.17.3323。PMC146090。_ PMID8811085。_ ^ Jurica MS、Monnat RJ、Stoddard BL(1998年10月)。「LAGLIDADGホーミングエンドヌクレアーゼI-CreIによるDNA認識と切断」。分子細胞。2(4):469–76。土井:10.1016 / s1097-2765(00)80146-x。PMID9809068。_ ^ セリグマン、LM; スティーブンス、KM; サベージ、JH; Monnat、RJ(1997)。「 EscherichiacoliにおけるクラミドモナスI- CreIモバイルイントロンホーミングシステムの遺伝子解析」。遺伝学。147(4):1653–1664。土井:10.1093/遺伝学/147.4.1653。PMC1208338。_ PMID9409828。_ ^ Heath PJ、Stephens KM、Monnat RJ、Stoddard BL(1997年6月)。「グループIイントロンにコードされたホーミングエンドヌクレアーゼであるI-Crelの構造」。自然構造生物学。4(6):468–76。土井:10.1038/nsb0697-468。PMID9187655。_ S2CID12261983。_ ^ Seligman LM、Chisholm KM、Chevalier BS、Chadsey MS、Edwards ST、Savage JH、Veillet AL。「ホーミングエンドヌクレアーゼの切断特異性を変化させる変異」。核酸研究。30(17):3870–9。土井:10.1093 / nar/gkf495。PMC137417。_ PMID12202772。_ ^ Sussman D、Chadsey M、Fauce S、Engel A、Bruett A、Monnat R、Stoddard BL、Seligman LM。「個々の標的部位の位置での新しいホーミングエンドヌクレアーゼ特異性の単離と特性化」。分子生物学ジャーナル。342(1):31–41。土井:10.1016/j.jmb.2004.07.031。PMID15313605。_ ^ Rosen LE、Morrison HA、Masri S、Brown MJ、Springstubb B、Sussman D、Stoddard BL、Seligman LM(2006)。「新規DNA標的特異性を有するホーミングエンドヌクレアーゼI-CreI誘導体」。核酸研究。34(17):4791–800。土井:10.1093 / nar/gkl645。PMC1635285。_ PMID16971456。_ ^ Arnould S、Chames P、Perez C、Lacroix E、Duclert A、Epinat JC、Stricher F、Petit AS、Patin A、Guillier S、Rolland S、Prieto J、Blanco FJ、Bravo J、Montoya G、Serrano L、Duchateau P、PâquesF。「新規DNA標的での組換えを誘導する多数の非常に特異的なホーミングエンドヌクレアーゼのエンジニアリング」。分子生物学ジャーナル。355(3):443–58。土井:10.1016/j.jmb.2005.10.065。PMID16310802。_ ^ Smith J、Grizot S、Arnould S、Duclert A、Epinat JC、Chames P、Prieto J、Redondo P、Blanco FJ、Bravo J、Montoya G、PâquesF、Duchateau P(2006)。「選択された配列を切断する人工ホーミングエンドヌクレアーゼを作成するためのコンビナトリアルアプローチ」。核酸研究。34(22):e149。土井:10.1093 / nar/gkl720。PMC1702487。_ PMID17130168。_ ^ Maggert KA、Golic KG。「ショウジョウバエのI-CreI発現によって生成される非常に効率的な性染色体交換」。遺伝学。171(3):1103–14。土井:10.1534/genetics.104.040071。PMC1456814。_ PMID16020774。_ “