Ribosomal_RNA
「RRNA」同社については、「ロールス・ロイス・ノース・アメリカ」を参照して
リボソームリボ核酸( rRNA ) は、リボソームの主成分であり、すべての細胞に不可欠な非コーディング RNAの一種です。rRNAはリボソーム内でタンパク質合成を行うリボザイムです。リボソーム RNA はリボソーム DNA ( rDNA) から転写され、リボソームタンパク質に結合して大小のリボソームサブユニットを形成します。rRNA は、トランスファー RNA (tRNA) とメッセンジャー RNA (mRNA) を強制的に処理してタンパク質に翻訳するリボソームの物理的および機械的因子です。リボソーム RNA は、ほとんどの細胞に見られる RNA の主な形式です。それ自体はタンパク質に翻訳されることはないにもかかわらず、細胞の RNA の約 80% を構成します。リボソームは、質量で約 60% の rRNA と 40% のリボソームタンパク質で構成されています。 rRNA さまざまな種のrRNA
識別子
その他のデータ
RNAの種類
遺伝子; rRNA
PDB構造
PDBe
コンテンツ
1 構造
1.1 組み立て
2 関数
3 サブユニットと関連するリボソーム RNA
3.1 原核生物では 3.2 真核生物では
4 生合成
4.1 真核生物では
4.1.1 真核生物の調節
4.2 原核生物では
4.2.1 原核生物の調節
5 劣化
5.1 真核生物では 5.2 原核生物では
6 配列の保存と安定性
7 意義
8 ヒトの遺伝子
9 こちらも参照
10 参考文献
11 外部リンク
構造
rRNA 配列の一次構造は生物によって異なりますが、これらの配列内の塩基対合は一般にステムループ構造を形成します。これらの rRNA ステムループの長さと位置により、種を超えて類似した 3 次元 rRNA 構造を作成できます。これらの構成により、rRNA はリボソームタンパク質と緊密で特異的な相互作用を形成し、リボソーム サブユニットを形成することができます。これらのリボソームタンパク質には、塩基性残基(酸性残基ではなく)と芳香族残基(すなわち、フェニルアラニン、チロシン、トリプトファン)が含まれており、スタッキング相互作用など、関連する RNA 領域と化学的相互作用を形成することができます。リボソームタンパク質は、塩基性残基 (つまり、リジンとアルギニン) からなる結合部位を使用して、rRNA の糖リン酸骨格に架橋結合することもできます。すべてのリボソームタンパク質 (rRNA に結合する特定の配列を含む) が同定されています。これらの相互作用と大小のリボソームサブユニットの結合により、タンパク質を合成できる機能するリボソームが生成されます。
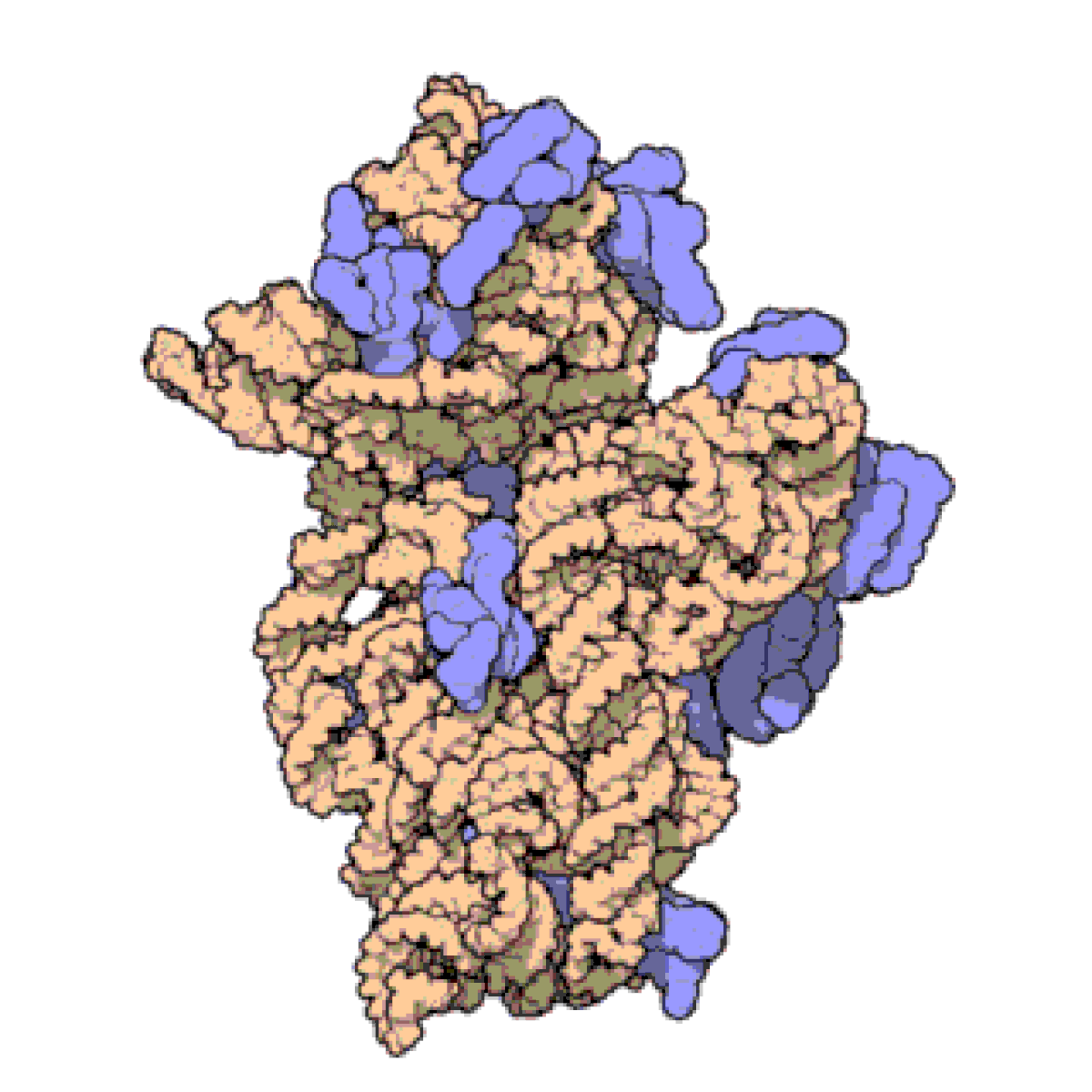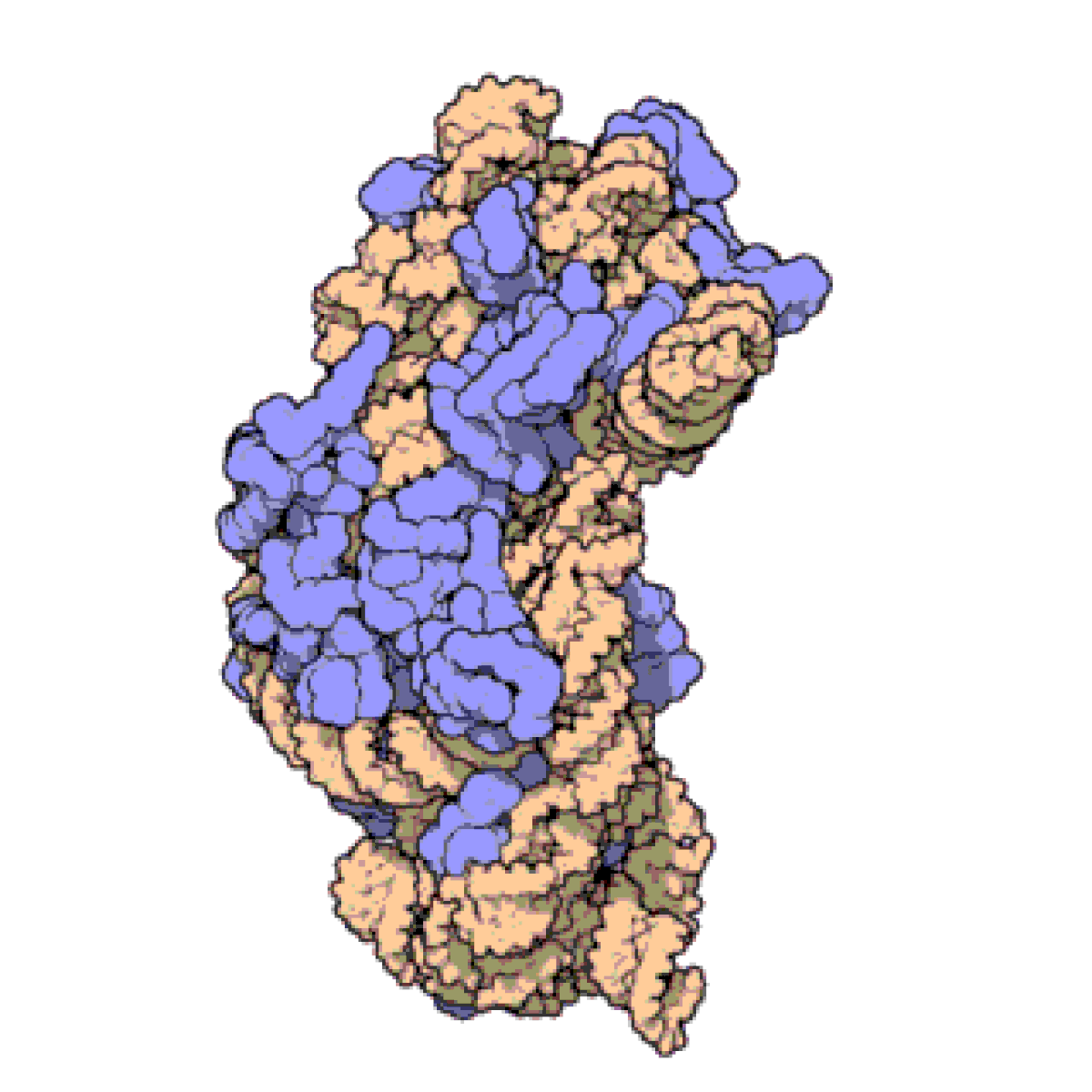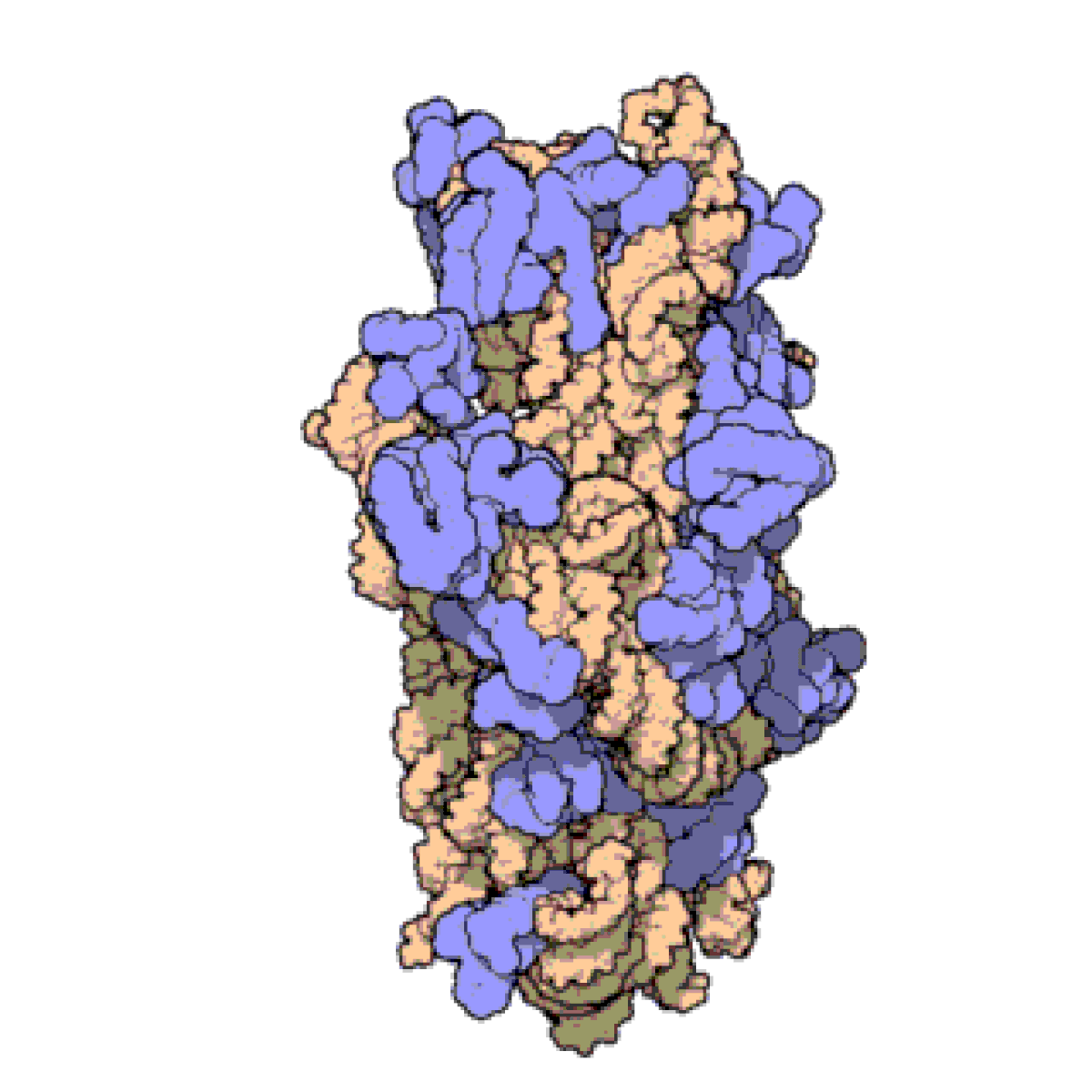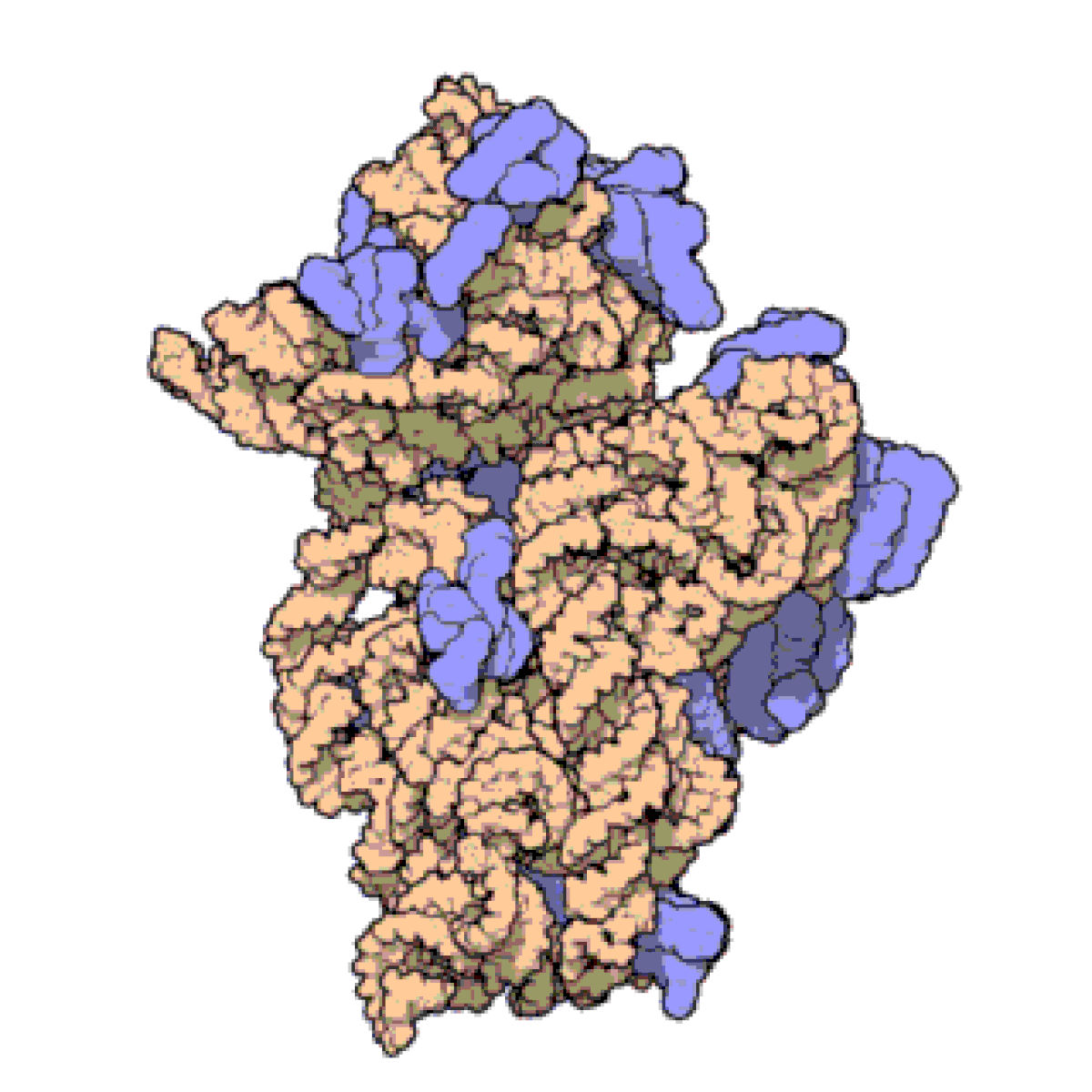
原核生物、特にサーマス・サーモフィラス
における完全に組み立てられたリボソーム RNA の小サブユニットの例。実際のリボソーム RNA (16S) はオレンジ色でコイル状に示されており、リボソームタンパク質は青色で結合しています。
リボソーム RNA は、大サブユニット (LSU) と小サブユニット (SSU) という 2 種類の主要なリボソーム サブユニットに構成されます。各タイプの 1 つが集まって、機能するリボソームを形成します。サブユニットは、サイズ沈降測定値 (接尾辞「S」の付いた数字) によって参照される場合が原核生物では、LSU と SSU はそれぞれ 50S サブユニットと 30S サブユニットと呼ばれます。真核生物では、それらは少し大きくなります。真核生物の LSU と SSU は、それぞれ 60S サブユニットと 40S サブユニットと呼ばれます。
細菌などの原核生物のリボソームでは、SSU には単一の小さな rRNA 分子 (約 1500 ヌクレオチド) が含まれ、LSU には単一の小さな rRNA 分子と単一の大きな rRNA 分子 (約 3000 ヌクレオチド) が含まれます。これらは約 50 個のリボソームタンパク質と結合してリボソーム サブユニットを形成します。原核生物のリボソームには 3 種類の rRNA が存在します。LSU には 23S および 5S rRNA、SSU には 16S rRNA が
ヒトなどの真核生物のリボソームでは、SSU には単一の小さな rRNA (約 1,800 ヌクレオチド) が含まれ、LSU には 2 つの小さな rRNA と 1 分子の大きな rRNA (約 5,000 ヌクレオチド) が含まれます。真核生物のrRNAには70を超えるリボソームタンパク質があり、それらが相互作用して原核生物と比較してより大きく、より多型のリボソームユニットを形成します。真核生物には 4 種類の rRNA があり、LSU に 3 種、SSU に 1 種です。 酵母は、真核生物のrRNA の挙動とプロセスを観察するための伝統的なモデルであり、研究の多様化に欠陥をもたらしてきました。技術の進歩 (特にクライオ EMの分野) により、他の真核生物におけるリボソームの挙動に関する予備調査が可能になったのは、ここ 10 年間のことです。酵母では、LSU に 5S、5.8S、および 28S rRNA が含まれています。5.8S と 28S を合わせたものは、真核生物でのみ発生すると考えられていたリボソームの表面に局在する拡張セグメント (ES) を除いて、サイズと機能が原核生物の 23S rRNA サブタイプとほぼ同等です。しかし最近、真核生物に最も近い古細菌の近縁種と考えられているアスガルド門、すなわちロキアーキオータとヘイムダラルキオータが、その 23S rRNA に 2 つの超大型 ES を保有していることが報告されました。同様に、5S rRNA には好塩性古細菌Halococcus morrhuaeのリボソームに 108 ヌクレオチドの挿入が含まれています。
真核生物の SSU には 18S rRNA サブユニットが含まれており、これには ES も含まれています。SSU ES は通常、LSU ES よりも小さいです。
SSU および LSU rRNA 配列は、古代の起源を持ち 、既知のすべての生命体に存在し、遺伝子の水平伝播に耐性があるため、生物間の進化的関係の研究に広く使用されています。rRNA 配列は、リボソームの機能における重要な役割により、時間が経っても保存されます (変化せず)。 16s rRNA に由来する系統情報は、現在、ヌクレオチドの類似性を計算することにより、類似した原核生物種を区別する主な方法として使用されています。正統な生命の樹は翻訳システムの系譜です。
LSU rRNA サブタイプは、リボソームタンパク質がこの領域のリボソームの触媒部位(特にペプチジルトランスフェラーゼセンター、つまり PTC)に結合できないため、リボザイムと呼ばれています。
SSU rRNA サブタイプは、その解読中心 (DC) で mRNA を解読します。リボソームタンパク質は DC に入ることができません。
rRNA の構造は、他の mRNA の翻訳中に、リボソームへの tRNA の結合に影響を与えるために劇的に変化する可能性が 16S rRNA では、これは、rRNA 内の特定のヌクレオチドが 1 つのヌクレオチドと別のヌクレオチドの間で塩基対を交互に配置し、rRNA の立体構造を変える「スイッチ」を形成するときに起こると考えられています。このプロセスはLSUとSSUの構造に影響を与える可能性があり、rRNA構造におけるこの構造スイッチが、tRNA選択においてコドンとアンチコドンを一致させる能力やmRNAを解読する能力においてリボソーム全体に影響を与えることを示唆しています。
組み立て
リボソーム RNA のリボソームへの組み込みと組み立ては、折り畳み、修飾、プロセシング、リボソームタンパク質との組み立てから始まり、2 つのリボソーム サブユニット、LSU と SSU を形成します。原核生物では、膜結合細胞小器官が欠如しているため、rRNA の取り込みが細胞質で起こります。しかし、真核生物では、このプロセスは主に核小体で起こり、プレ RNA の合成によって開始されます。これには 3 つの RNA ポリメラーゼがすべて存在する必要が実際、RNA ポリメラーゼ I によるプレ RNA の転写は、細胞の全細胞 RNA 転写の約 60% を占めます。これに続いて、プレ RNA がリボソームタンパク質と組み立てられるように折りたたまれます。この折り畳みは、エンドヌクレアーゼおよびエキソヌクレアーゼ、RNAヘリカーゼ、GTPase、ATPaseによって触媒されます。その後、rRNA はエンドおよびエキソヌクレオチド分解プロセシングを受けて、外部および内部の転写されたスペーサーを除去します。次に、プレ RNA は、リボソーム構築因子とリボソームタンパク質がプレ RNA と集合してプレリボソーム粒子を形成する前に、メチル化やシュードウリジニル化などの修飾を受けます。さらなる成熟段階を経て核小体から細胞質へ出ると、これらの粒子は結合してリボソームを形成します。 rRNA の一次構造内に見られる塩基性残基と芳香族残基は、好ましいスタッキング相互作用とリボソームタンパク質への誘引を可能にし、rRNA の骨格とリボソーム単位の他の構成要素との間に架橋効果を生み出します。これらのプロセスの開始と開始部分の詳細については、「生合成」セクションを参照して
関数
リボソームの簡略化した図 (ここでは視覚化の目的で SSU と LSU が人工的に分離されています)。A サイトと P サイト、および連携して動作する小型および大型リボソーム サブユニットの両方を示しています。
さまざまな種の間で rRNA の二次構造要素が普遍的に保存されていることから、これらの配列は発見された最も古い配列の一部であることが示されています。それらは、mRNA の翻訳の触媒部位の形成において重要な役割を果たします。mRNAの翻訳中、rRNAはmRNAとtRNAの両方に結合して、mRNAのコドン配列をアミノ酸に翻訳するプロセスを促進するように機能します。tRNAがSSUとLSUの間に挟まれると、rRNAがタンパク質合成の触媒作用を開始します。SSU では、mRNA は tRNA のアンチコドンと相互作用します。LSU では、tRNA のアミノ酸アクセプター ステムが LSU rRNA と相互作用します。リボソームはエステルとアミドの交換を触媒し、新生ペプチドの C 末端を tRNA からアミノ酸のアミンに転移します。これらのプロセスは、rRNA ステムループによって形成される、これらの分子が結合できるリボソーム内の部位によって発生する可能性がリボソームには、A、P、E サイトと呼ばれる結合部位が 3 つ
一般に、A (アミノアシル) 部位には、アミノアシル tRNA ( 3′ 末端のアミノ酸がエステル化されたtRNA ) が含まれています。
P (ペプチジル) 部位には、新生ペプチドにエステル化されたtRNAが含まれています。 A 部位tRNAの遊離アミノ (NH 2 ) 基は、P 部位 tRNA のエステル結合を攻撃し、新生ペプチドの A 部位のアミノ酸への転移を引き起こします。この反応はペプチジルトランスフェラーゼ中心で起こります
E (出口) 部位には、遊離 3′ 末端 (アミノ酸や新生ペプチドを持たない) を備えた、放出されたtRNAが含まれています。
単一のmRNA は、複数のリボソームによって同時に翻訳されます。これはポリソームと呼ばれます。
原核生物では、 mRNAの翻訳における rRNA の重要性をさらに特定するために多くの研究が行われてきました。例えば、A 部位は主に 16S rRNA から構成されていることがわかっています。この部位でtRNAと相互作用するさまざまなタンパク質要素とは別に、これらのタンパク質がリボソーム構造を変えることなく除去された場合、その部位は正常に機能し続けるだろうという仮説が立てられています。P 部位では、結晶構造の観察により、 16s rRNA の 3′ 末端がmRNAの分子のようにこの部位に折り畳まれることが示されています。これにより、サブユニットを安定化する分子間相互作用が生じます。同様に、A 部位と同様に、P 部位には主に rRNA が含まれ、タンパク質はほとんど含まれません。たとえば、ペプチジルトランスフェラーゼ中心は、23S rRNA サブユニットのヌクレオチドによって形成されます。実際、研究により、ペプチジルトランスフェラーゼ中心にはタンパク質が含まれておらず、完全に rRNA の存在によって開始されることが示されています。A サイトや P サイトとは異なり、E サイトにはより多くのタンパク質が含まれています。タンパク質はA および P サイトの機能に必須ではないため、E サイトの分子組成は、おそらく後で進化したことを示しています。原始的なリボソームでは、 tRNA がP 部位から出た可能性がさらに、E 部位tRNA は16S および 23S rRNA サブユニットの両方と結合することが示されています。
サブユニットと関連するリボソーム RNA
リボソーム RNA の種類と、それらがどのように結合してリボソーム サブユニットを作成するかを示す図。
原核生物と真核生物のリボソームは両方とも、大きいサブユニットと小さいサブユニットの 2 つのサブユニットに分解できます。それぞれの rRNA について以下の表で使用されている例示的な種は、細菌Escherichia coli (原核生物) とヒト (真核生物) です。「nt」はヌクレオチド単位でのrRNAタイプの長さを表し、「S」(「16S」など)はスヴェドベリ単位を表すことに注意して
タイプ サイズ 大サブユニット ( LSU rRNA ) 小サブユニット ( SSU rRNA )
原核生物
70年代
50S(5S :120nt、23S :2906nt)
30S(16S :1542nt)
真核生物(核)
80年代
60S ( 5S : 121 nt、 5.8S : 156 nt、 28S : 5070 nt )
40S ( 18S : 1869 nt )
真核生物(ミトコンドリア) 55S 39S 16S (ミトコンドリアにコードされた 16S rRNA : 約 1,571 nt)
28S 12S (ミトコンドリアにコードされた 12S rRNA : 約 955 nt)
サブユニットの S ユニット (または rRNA) は、質量ではなく沈降速度の尺度を表すため、単純に追加することはできません。各サブユニットの沈降速度は、その質量だけでなくその形状にも影響されます。nt ユニットは、直鎖状 rRNA ポリマーのユニットの整数を表すため、追加できます (たとえば、ヒト rRNA の全長 = 7216 nt)。
rRNAをコードする遺伝子クラスターは一般に「リボソームDNA」またはrDNAと呼ばれます(この用語はリボソームにDNAが含まれていると暗示しているように思われますが、実際はそうではないことに注意してください)。
原核生物では
原核生物では、小さな 30S リボソーム サブユニットに16S リボソーム RNAが含まれています。大きな 50S リボソーム サブユニットには 2 つの rRNA 種 (5S および23S リボソーム RNA ) が含まれています。したがって、細菌と古細菌の両方に、16S、23S、および 5S の 3 つの rRNA タイプすべてをコードする 1 つの rRNA 遺伝子が存在すると推測できます。
細菌の 16S リボソーム RNA、23S リボソーム RNA、および 5S rRNA 遺伝子は、通常、共転写オペロンとして組織されます。このセクションの画像に示されているように、16S と 23S rRNA遺伝子の間には内部転写スペーサーがオペロンのコピーが 1 つ以上、ゲノム中に分散している可能性があります(たとえば、大腸菌には 7 つあります)。通常、細菌には 1 ~ 15 個のコピーが存在します。
古細菌には、単一の rRNA 遺伝子オペロン、または同じオペロンの最大 4 コピーのいずれかが含まれます。
16S リボソーム RNA (リボソーム内) の 3′ 末端は、シャイン-ダルガーノ配列と呼ばれるmRNAの 5′ 末端の配列を認識します。
真核生物では
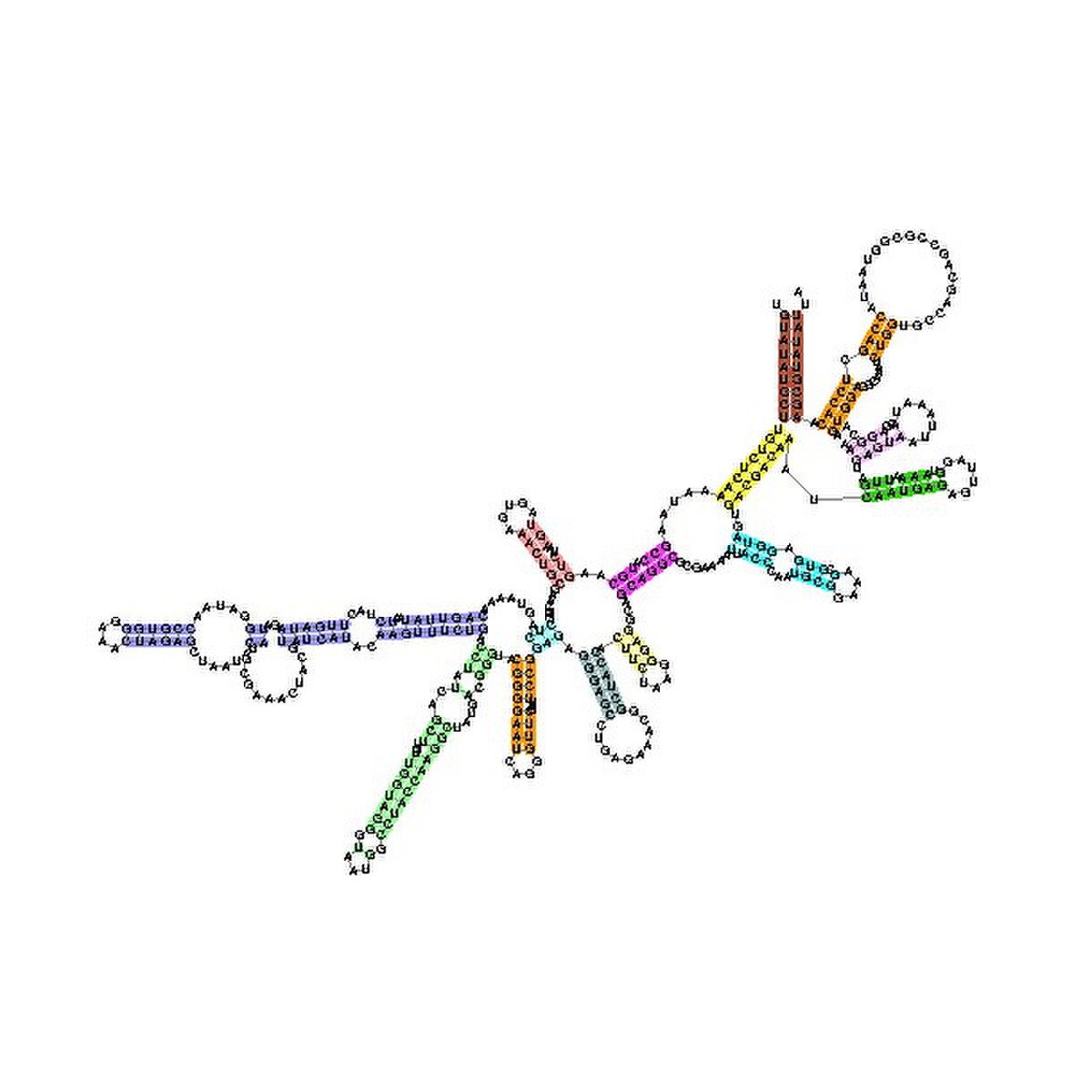
小サブユニット リボソーム RNA、5′ ドメインはRfamデータベースから取得。この例はRF00177で、培養されていない細菌からのフラグメントです。
対照的に、真核生物は一般に、タンデムリピートで組織化されたrRNA遺伝子のコピーを多数持っています。ヒトでは、約 300 ~ 400 のリピートが 5 つのクラスターに存在し、染色体13 ( RNR1 )、14 ( RNR2 )、15 ( RNR3 )、21 ( RNR4 )、および 22 ( RNR5 ) に位置します。二倍体ヒトは 10 個のゲノムrDNAクラスターを持ち、合計でヒトゲノムの 0.5% 未満を構成します。
以前は、リピートrDNA配列は同一であり、自然な複製エラーや点突然変異を説明するための冗長性またはフェイルセーフとして機能することが認められていました。しかし、ヒトのrDNA (およびその後の rRNA) の配列変動は、複数の染色体にわたって、ヒト個体内およびヒト個体間で観察されています。これらのバリエーションの多くは回文配列であり、複製による潜在的なエラーです。特定の変異体はマウスでも組織特異的に発現します。
哺乳類細胞は、2 つのミトコンドリア ( 12Sおよび16S ) rRNA 分子と 4 種類の細胞質 rRNA (28S、5.8S、18S、および 5S サブユニット) を持っています。28S、5.8S、および 18S rRNA は、2 つの内部転写スペーサーによって分離された単一の転写単位 (45S) によってコードされます。最初のスペーサーは細菌や古細菌に見られるものに対応し、もう 1 つのスペーサーは原核生物の 23S rRNA に挿入されたものです。 45S rDNA は、染色体 13、14、15、21、および 22 上の 5 つのクラスター (それぞれ 30 ~ 40 のリピートを持つ) に編成されています。これらはRNA ポリメラーゼ Iによって転写されます。5S サブユニットの DNA はタンデム配列(約 200 ~ 300 個の真の 5S 遺伝子と分散した多くの偽遺伝子) で存在し、染色体 1q41 ~ 42 上に最大のものです。5S rRNA はRNA ポリメラーゼ IIIによって転写されます。ほとんどの真核生物の18S rRNAはリボソームの小サブユニットにあり、大サブユニットには 3 つの rRNA 種 (哺乳類の5S、5.8S、および28S、植物の 25S、rRNA) が含まれています。
ハエでは、大サブユニットには 3 つではなく 4 つの rRNA 種が含まれており、5.8S rRNA に分割があり、より短い 5.8S サブユニット (123 nt) と 2S rRNA と呼ばれる 30 ヌクレオチドのサブユニットが存在します。両方のフラグメントは、内部転写された 28 ヌクレオチドのスペーサーによって分離されています。2S rRNA は小さく、非常に豊富であるため、その存在は sRNA ライブラリの構築を妨げ、他の sRNA の定量を損なう可能性が2S サブユニットはショウジョウバエやキノコのブヨ種では検出されますが、蚊には存在しません。
小サブユニットリボソーム RNA (SSU rRNA) の三次構造は、X 線結晶構造解析によって解明されました。 SSU rRNA の二次構造には、5’、中央、3′ メジャー、および 3′ マイナー ドメインの 4 つの異なるドメインが含まれています。5′ ドメインの二次構造のモデル(500 ~ 800ヌクレオチド) が示されています。
生合成
詳細は「リボソームの生合成」を参照
真核生物では
細胞小器官の構成要素であるrRNA の生成は、最終的にはリボソーム合成の律速段階となります。核小体では、rRNA は、ゲノム全体に繰り返し存在する、それをコードする特殊遺伝子 ( rDNA ) を使用して、RNA ポリメラーゼ Iによって合成されます。 18S、28S、および 5.8S rRNA をコードする遺伝子は核小体オーガナイザー領域に位置し、 RNA ポリメラーゼ Iによって大きな前駆体 rRNA (pre-rRNA) 分子に転写されます。これらの pre-rRNA 分子は外部および内部のスペーサー配列によって分離され、その後メチル化されます。これは、後の組み立てとフォールディングの鍵となります。 個々の分子として分離および放出された後、アセンブリタンパク質は裸の各 rRNA 鎖に結合し、協調的なアセンブリと必要に応じてさらに多くのフォールディングタンパク質の漸進的な追加を使用して、それを機能的な形態に折り畳みます。フォールディングタンパク質がどのようにrRNAに結合するのか、またどのようにして正しいフォールディングが達成されるのかについての正確な詳細は依然として不明である。その後、rRNA 複合体は、タンパク質と複合体を形成したsnoRNA (核小体低分子 RNA)によって誘導されるエキソおよびエンドヌクレオチド切断を伴う反応によってさらに処理されます。これらの複合体が圧縮されて粘着ユニットを形成すると、rRNA と周囲のリボソームタンパク質の間の相互作用は、安定性を提供し、結合部位を保護するために、構築中に常に再構築されます。このプロセスは、rRNA ライフサイクルの「成熟」段階と呼ばれます。rRNA の成熟中に起こる修飾は、 tRNAとmRNAの翻訳アクセスの物理的調節を提供することにより、遺伝子発現の制御に直接寄与することがわかっています。いくつかの研究では、リボソームの安定性を維持するには、この期間中にさまざまな rRNA タイプの大規模なメチル化も必要であることが判明しました。
5S rRNA の遺伝子は核小体の内部に位置し、 RNA ポリメラーゼ IIIによってプレ 5S rRNA に転写されます。 pre-5S rRNA は核小体に入り、28S および 5.8S rRNA とプロセシングおよび集合して LSU を形成します。18S rRNA は、多数のリボソームタンパク質と結合して SSU を形成します。両方のサブユニットが組み立てられると、それらは個別に細胞質に輸送されて80S ユニットを形成し、mRNAの翻訳が開始されます。
リボソーム RNA は非コーディングであり、いかなる種類のタンパク質にも翻訳されることはありません。rRNA はrDNAから転写されるだけで、その後、リボソームの構造構築ブロックとして使用するために成熟します。転写された rRNA はリボソームタンパク質に結合してリボソームのサブユニットを形成し、 mRNAとtRNA をリボソームに押し出して処理および翻訳する物理的構造として機能します。
真核生物の調節
rRNA の合成は、さまざまなプロセスと相互作用によって恒常性を維持するために上方制御および下方制御されます。
RNAポリメラーゼ I は AKT 依存性であるため、キナーゼAKT は間接的に rRNA の合成を促進します 。
アンギオゲニン(ANG)などの特定の血管新生リボヌクレアーゼは、核小体に移動して蓄積する可能性がANG の濃度が高すぎると、ANG がrDNAのプロモーター領域に結合し、rRNA の転写を不必要に増加させる可能性があることがいくつかの研究でわかっています。これは核小体に損傷を与える可能性があり、チェックされない転写や癌を引き起こす可能性さえ
細胞のグルコース制限中、AMP 活性化プロテインキナーゼ(AMPK) は、エネルギーを消費するが必須ではない代謝プロセスを妨げます。その結果、 RNA ポリメラーゼ I (Ser-635 部位) をリン酸化して、転写開始を妨害することで rRNA 合成を下方制御することができます。
リボソーム解読中心から複数のプソイドウリジンまたは 29-O-メチル化領域を損傷または除去すると、新しいアミノ酸の取り込み速度が低下するため、rRNA転写速度が大幅に低下します。
ヘテロクロマチンの形成はrRNA 転写をサイレンシングするために不可欠であり、ヘテロクロマチンの形成がないとリボソーム RNA が無制限に合成され、生物の寿命が大幅に短縮されます。
原核生物では
真核生物と同様に、rRNA の生成は、原核生物のリボソーム合成における律速段階です。大腸菌では、 rRNA が 7 つの異なるrrnオペロン内にある 2 つのプロモーター P1 および P2 から転写されることが判明しています。P1プロモーターは、中程度から高い細菌増殖速度の間の rRNA 合成の制御に特に関与しています。このプロモーターの転写活性は増殖速度に直接比例するため、主に rRNA制御に関与します。rRNA 濃度の増加は、リボソーム合成に対する負のフィードバック機構として機能します。rrn P1 プロモーターの効率的な転写には、高い NTP 濃度が必要であることがわかっています。これらは、RNA ポリメラーゼおよびプロモーターと安定化複合体を形成すると考えられています。特に細菌では、高 NTP 濃度と rRNA 合成の増加との関連性が、なぜリボソーム合成、つまりタンパク質合成が増殖速度に依存するのかについて分子的な説明を提供します。増殖速度が低いと、rRNA / リボソーム合成速度が低くなり、増殖速度が高いと、rRNA / リボソーム合成速度が高くなります。これにより、細胞は、そのニーズと利用可能なリソースに応じて、エネルギーを節約したり、代謝活動を増加したりすることができます。
原核細胞では、各 rRNA 遺伝子またはオペロンは、転写されたスペーサーとともに16S、23S、5S rRNA およびtRNA配列を含む単一の RNA 前駆体に転写されます。その後、転写が完了する前に RNA のプロセシングが始まります。プロセシング反応中に、rRNA とtRNA は別々の分子として放出されます。
原核生物の調節
rRNA は原核生物の細胞生理において重要な役割を果たしているため、rRNA制御機構には多くの重複が転写レベルでは、細胞の恒常性の維持を促進する rRNA 転写の正のエフェクターと負のエフェクターの両方が存在します。
rrn P1 プロモーターの上流にある UP エレメントは、RNA ポリメラーゼのサブユニットに結合することができ、rRNA の転写を促進します。
FIS などの転写因子はプロモーターの上流に結合し、転写を促進するRNA ポリメラーゼと相互作用します。
抗終結因子は、rrn P2プロモーターの下流に結合し、早期の転写終結を防ぎます。
厳密な応答により、アミノ酸の利用可能性が低い場合、ppGpp (ネガティブエフェクター) はP1 および P2プロモーターの両方からの転写を阻害する可能性が
劣化
リボソーム RNA は、他の一般的なタイプの RNA と比較して非常に安定しており、健康な細胞環境で長期間存続します。リボソーム内のリボソーム RNA は、一度機能単位に組み立てられると、細胞の生活環の定常期において何時間も安定しています。分解は、リボソームの「失速」によって引き起こされる可能性があり、この状態は、リボソームが欠陥のある mRNA を認識するか、リボソームによる翻訳の停止を引き起こすその他の処理上の困難に遭遇したときに発生します。リボソームが停止すると、リボソーム上の特殊な経路が開始され、複合体全体が分解の対象となります。
真核生物では
他のタンパク質やRNAと同様、rRNA の生成にはエラーが発生しやすく、その結果、機能しない rRNA が生成されます。これを修正するために、細胞は非機能的 rRNA 分解 (NRD) 経路による rRNA の分解を可能にします。このトピックの研究の多くは、真核細胞、特にサッカロマイセス・セレビシエ酵母に対して行われました。現在、真核生物において細胞がどのようにして機能的に欠陥のあるリボソームを標的にしてユビキチン化と分解を行うことができるのかについては、基本的な理解しか得られていない。
40S サブユニットの NRD 経路は、60S サブユニットの NRD 経路から独立していても、別個であってもよい。特定の遺伝子は特定のプレ RNA の分解に影響を与えることができますが、他の遺伝子には影響を及ぼさないことが観察されています。
Mms1p や Rtt101p など、多数のタンパク質が NRD 経路に関与しており、これらは複合体を形成してリボソームを分解の標的にすると考えられています。Mms1p と Rtt101p は結合することがわかっており、Rtt101p はユビキチンE3リガーゼ複合体を動員し、機能しないリボソームが分解される前にユビキチン化されることを可能にすると考えられています。
原核生物には Mms1 のホモログが存在しないため、原核生物が機能しない rRNA をどのように分解できるのかは不明です。
真核細胞の増殖速度は、機能しない rRNA の蓄積によって有意な影響を受けることはないようです。
原核生物では
真核生物と比較して、原核生物におけるリボソーム RNA 分解に関する利用可能な研究ははるかに少ないですが、細菌が真核生物の NRD と比較して同様の分解スキームに従うかどうかについては依然として関心が寄せられています。原核生物に関して行われた研究の多くは大腸菌に対して行われています。真核生物と原核生物のrRNA分解には多くの違いが見つかり、研究者らは両者は異なる経路で分解すると考えている。
真核生物では rRNA 分解を引き起こすことができた rRNA の特定の変異は、原核生物では分解を引き起こすことができませんでした。
1 つのサブユニットの変異によってそのサブユニットのみが分解される真核生物と比較して、23S rRNA の点変異は23S および 16S rRNAの両方の分解を引き起こします。
研究者らは、23S rRNA からヘリックス構造全体 (H69) を除去しても、その分解が引き起こされないことを発見しました。このことから、エンドヌクレアーゼが変異した rRNA を認識して分解するには H69 が不可欠であると研究者らは考えました。
配列の保存と安定性
rRNA はあらゆる生物体に広く普及し、揺るぎない性質を持っているため、生物体を破壊することなく、遺伝子導入、突然変異、改変に対する rRNA の耐性を研究することが、人気の高い分野となっています。リボソーム RNA 遺伝子は修飾や侵入に対して耐性があることがわかっています。rRNA の配列が変化すると、細胞が損なわれ、すぐに正常な機能を停止することがわかっています。 rRNA のこれらの重要な特性は、さまざまな生物学的ドメインにわたるリボソーム RNA 配列のアライメントによって「分類学的割り当て」が大幅に容易になる遺伝子データベース プロジェクト (SILVA や SINA などの包括的なオンライン リソース) にとって特に重要になっています。系統解析と微生物の多様性の調査。」
回復力の例:
16S rRNA ユニットの多くの部分に大きくて意味のない RNA フラグメントを追加しても、全体としてリボソームユニットの機能が変化することは観察されません。
ノンコーディング RNA RD7 は、 rRNA のプロセシングを変更して分子をカルボン酸による分解に耐性のあるものにする能力を持っています。これは、酸の蓄積(ATP生成に必要な基質のリン酸化による)が細胞内機能にとって有毒になる可能性がある活発な増殖中に、rRNA 濃度を維持する上で重要なメカニズムです。
16S rRNA に沿ってシス切断できるハンマーヘッド リボザイムを挿入すると、機能が大幅に阻害され、安定性が低下します。
ほとんどの細胞機能は、低酸素環境に短期間さらされただけで大きく低下しますが、rRNA は 6 日間の長期低酸素状態の後でも分解されずに解決されます。このような長期間が経過した後にのみ、rRNA 中間体 (最終的に分解が起こっていることを示します) が存在し始めます。
意義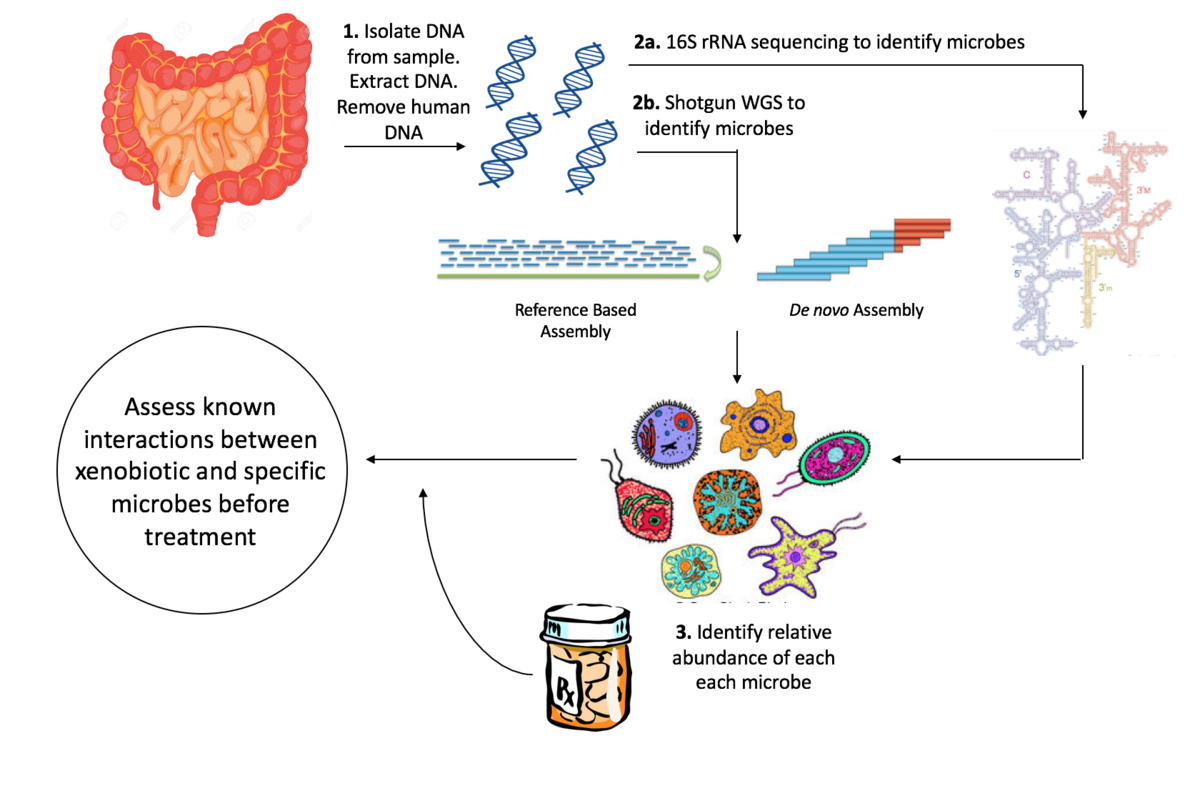
この図は、原核生物における rRNA 配列決定を最終的にどのように使用して、rRNA が最初に得られた微生物によって引き起こされる病気と戦うための医薬品を製造できるかを示しています。
リボソーム RNA の特徴は進化、ひいては分類学と医学において重要です。
rRNA は、すべての細胞に存在する数少ない遺伝子産物の 1 つです。このため、rRNA ( rDNA )をコードする遺伝子は、生物の分類群を特定し、関連する群を計算し、種の分岐率を推定するために配列決定されます。その結果、何千もの rRNA 配列が知られており、RDP-II や SILVA などの特殊なデータベースに保存されています。
rRNA の変化により、結核菌(結核を引き起こす細菌)などの特定の病気の原因となる細菌が極度の薬剤耐性を発現することが可能になります。同様の問題により、これは獣医学でも一般的な問題となっており、ペットの細菌感染症に対処する主な方法は、細菌のリボソームのペプチジルトランスフェラーゼセンター(PTC)を攻撃する薬剤の投与である。23S rRNA の変異は、これらの薬剤が未知の方法で連携して PTC を完全に回避するため、これらの薬剤に対する完全な耐性を生み出しました。
rRNA は、クロラムフェニコール、エリスロマイシン、カスガマイシン、ミクロコックシン、パロモマイシン、リシン、α-サルシン、スペクチノマイシン、ストレプトマイシン、チオストレプトンなど、臨床的に関連する数多くの抗生物質の標的です。
rRNA は、ヒトのmiR-663やマウスのmiR-712など、種特異的なマイクロ RNAの起源であることが示されています。これらの特定のmiRNA は、 rRNA の内部転写スペーサーに由来します。
ヒトの遺伝子
45S: RNR1、RNR2、 RNR3、RNR4、RNR5 ; (クラスター化されていない) RNA18SN1 、RNA18SN2 、 RNA18SN3 、 RNA18SN4 、 RNA18SN5 、 RNA28SN1 、RNA28SN2、RNA28SN3、RNA28SN4、RNA28SN5、RNA45SN1、RNA45SN2、RNA45SN3 、 RNA45SN4 、RNA45 SN5 、RNA5-8SN1 、 RNA5-8SN2、RNA5-8SN3、RNA5-8SN4、RNA5 -8SN5
5S: RNA5S1、RNA5S2、RNA5S3、RNA5S4、RNA5S5、RNA5S6、RNA5S7、RNA5S8、RNA5S9、RNA5S10、RNA5S11、RNA5S12、RNA5S13、RNA5S14、RNA5S15、RNA5S16 、 RNA5S17
Mt:MT-RNR1、MT-TV(共用)、MT-RNR2
こちらも参照
リボタイピング
ジアザボリン B、リボソーム大サブユニットの rRNA の成熟阻害剤
参考文献
^ バーク、アーノルド; ボルチモア、デイビッド。ロディッシュ、ハーベイ。ダーネル、ジェームズ。松平、ポール。ジプルスキー、S. ローレンス (1996-01-31)。モレクラレ・ゼルバイオロジー。ベルリン、ボストン:DE GRUYTER。土井: 10.1515/9783110810578。ISBN 9783110810578。
^ ロディッシュ、ハーベイ; バーク、アーノルド。ジプルスキー、S.ローレンス。松平、ポール。ボルチモア、デイビッド。ダーネル、ジェームス (2000)。「タンパク質合成における RNA の 3 つの役割」。分子細胞生物学。第 4 版。
^ ウルラウブ H、クルフト V、ビショフ O、ミュラー EC、ヴィットマン=リーボルド B (1995 年 9月)。「架橋研究によって決定されたリボソームにおけるタンパク質-rRNA結合の特徴とその構造的および機能的影響」。EMBO ジャーナル。14 (18): 4578–88。土井:10.1002/j.1460-2075.1995.tb00137.x。PMC 394550。PMID 7556101。 ^ Ferreira-Cerca S、Pöll G、Gleizes PE、Tschochner H、Milkereit P 。「プレ 18S rRNA の成熟と輸送、およびリボソーム機能における真核生物のリボソームタンパク質の役割」。分子細胞。20 (2): 263-75。土井:10.1016/j.molcel.2005.09.005。PMID 16246728。 ^ Szymański M、Barciszewska MZ、Erdmann VA、Barciszewski J 。「5 S rRNA: 構造と相互作用」。生化学ジャーナル。371 (Pt 3): 641–51。土井:10.1042/bj20020872。PMC 1223345。PMID 12564956。 ^ ヘンラス AK、プリッソン=チャスタン C、オドノヒュー MF、チャクラボルティ A、グレイズ PE (2015-03-01)。「真核生物におけるプレリボソームRNAプロセシングの概要」。Wiley Interdisciplinary Reviews: RNA。6 (2): 225-42。土井:10.1002/wrna.1269。PMC 4361047。PMID 25346433。 ^ ペネフ PI、ファクレタハ=アヴァル S、パテル VJ、カンノーネ JJ、グテル RR、ペトロフ AS、ウィリアムズ LD、グラス JB 。「アスガルド古細菌における超大型リボソーム RNA 伸長セグメント」。ゲノム生物学と進化。12 (10): 1694 ~ 1710 年。土井:10.1093/gbe/evaa170。PMC 7594248。PMID 32785681。 ^ ルーセン、KR。デラウェア州ニコルソン。ワシントンDC、ユーバンクス。ジョージア州フォックス(1981年5月)。「古細菌の 5S rRNA には長い挿入配列が含まれています。」自然。293 (5835): 755–756。Bibcode : 1981Natur.293..755L。土井: 10.1038/293755a0。PMID 6169998。S2CID 4341755。 ^ ティルマライ、MR; JT ケルバー; パーク、DR; トラン、Q; ジョージア州フォックス(2020年8月31日)。「極好塩性古細菌Halococcus morrhuaeの 5S リボソーム RNA における大きな挿入の低温電子顕微鏡による可視化」。FEBS オープンバイオ。10 (10): 1938 ~ 1946 年。土井: 10.1002/2211-5463.12962。PMC 7530397。PMID 32865340。 ^ Woese CR、Fox GE (1977 年 11月)。「原核生物ドメインの系統学的構造: 主要な界」。手順 国立 アカド。科学。アメリカ。74 (11): 5088–5090。Bibcode : 1977PNAS…74.5088W。土井: 10.1073/pnas.74.11.5088。PMC 432104。PMID 270744。 ^ Lagesen K、Hallin P、Rødland EA、Staerfeldt HH、Rognes T、Ussery DW (2007-05-01)。「RNAmmer: リボソーム RNA 遺伝子の一貫した迅速なアノテーション」。核酸研究。35 (9): 3100–8。土井:10.1093/nar/gkm160。PMC 1888812。PMID 17452365。 ^ Chun J、Lee JH、Jung Y、Kim M、Kim S、Kim BK、Lim YW 。「EzTaxon: 16S リボソーム RNA 遺伝子配列に基づいて原核生物を同定するための Web ベースのツール」。系統的および進化的微生物学の国際ジャーナル。57 (Pt 10): 2259–61。土井:10.1099/ijs.0.64915-0。PMID 17911292。 ^ ティルマライ MR、リバス M、トラン Q、フォックス GE 。「ペプチジルトランスフェラーゼセンター:過去への窓」。微生物 Mol Biol Rev . 85 (4): e0010421。土井:10.1128/MMBR.00104-21。PMC 8579967。PMID 34756086。 ^ ゴーシュ、アルナブ; コマール、アントン A (2015 年 1 月 2 日) 「小サブユニットのリボソームタンパク質における真核生物特異的拡張: 構造と機能」。翻訳。3 (1): e999576。土井: 10.1080/21690731.2014.999576。PMC 4682806。PMID 26779416。 ^ ロドメル JS、ダールバーグ AE (1997 年 8月)。「メッセンジャーRNAの解読中の大腸菌16SリボソームRNAの立体構造スイッチ」。科学。277 (5330): 1262–7。土井:10.1126/science.277.5330.1262。PMID 9271564。 ^ ガバシュビリ IS、アグラワル RK、グラスッチ R、スクワイアズ CL、ダールバーグ AE、フランク J (1999 年 11月)。「16S リボソーム RNA の立体構造スイッチによって引き起こされる 70S リボソーム 3D 構造の主要な再構成」。EMBO ジャーナル。18 (22): 6501–7。土井: 10.1093/emboj/18.22.6501。PMC 1171713。PMID 10562562。 ^ ウールフォード JL、バゼルガ SJ 。「酵母サッカロミセス・セレビシエにおけるリボソーム生合成」。遺伝学。195 (3): 643–81。土井:10.1534/genetics.113.153197。PMC 3813855。PMID 24190922。
^ Baßler J、Hurt E . 「真核生物のリボソーム集合体」。生化学の年次レビュー。88 (1): 281–306。土井: 10.1146/annurev-biochem-013118-110817。PMID 30566372。S2CID 58650367。 ^ ムーア PB、シュタイツ TA 。「リボソーム機能におけるRNAの関与」。自然。418 (6894): 229–35。Bibcode : 2002Natur.418..229M。土井:10.1038/418229a。PMID 12110899。S2CID 4324362。 ^ “ホモ・サピエンス5S リボソーム RNA” . 2018年5月24日。
^ “ホモ・サピエンス5.8S リボソーム RNA” . 2017年2月10日。
^ “ホモ・サピエンス28S リボソーム RNA” . 2017年2月4日。
^ “ホモ・サピエンスの18SリボソームRNA” . 2017年2月4日。
^ カウシャル、PS; シャルマ氏、MR。英国アグラワル。「55S 哺乳動物のミトコンドリア リボソームとその tRNA 出口領域」。ビオキミー。114:119-26。土井:10.1016/j.biochi.2015.03.013。PMC 4772884。PMID 25797916。 ^ ストッダード SF、スミス BJ、ハイン R、ローラー BR、シュミット TM 。「rrnDB: 細菌および古細菌における rRNA 遺伝子量を解釈するための改良されたツールと、将来の開発のための新しい基盤」。核酸研究。43 (データベースの問題): D593-8。土井:10.1093/nar/gku1201。PMC 4383981。PMID 25414355。
^ Lafontaine DL、Tollervey D 。「リボソームの機能と合成」。Nature は分子細胞生物学をレビューします。2 (7):514-20。土井:10.1038/35080045。hdl : 1842/729。PMID 11433365。S2CID 2637106。 ^ Stults DM、Killen MW、Williamson EP、Hourigan JS、Vargas HD、Arnold SM、他。。「ヒトrRNA遺伝子クラスターは癌における組換えホットスポットである」。がん研究。69 (23): 9096–104。土井: 10.1158/0008-5472.can-09-2680。PMID 19920195。S2CID 6162867。 ^ Kim JH、Dilthey AT、Nagaraja R、Lee HS、Koren S、Dudekula D、他。。「TAR クローニングとロングリード配列決定によって特徴付けられるヒト 21 番染色体リボソーム RNA 遺伝子の変異」。核酸研究。46 (13): 6712–6725。土井:10.1093/nar/gky442。PMC 6061828。PMID 29788454。 ^ パークスMM、キュリロCM、ダスRA、ボイマーL、ライデンD、ヴィンセントCT、ブランチャードSC 。「変異型リボソーム RNA 対立遺伝子は保存されており、組織特異的な発現を示します。 」科学の進歩。4 (2):eaao0665。Bibcode : 2018SciA….4..665P。土井:10.1126/sciadv.aao0665。PMC 5829973。PMID 29503865。 ^ 島田哲也 (1992 年 8月)。「双翅目における分割 5.8S リボソーム RNA の分布」。昆虫の分子生物学。1 (1): 45-48。土井:10.1111/j.1365-2583.1993.tb00076.x。ISSN 0962-1075。PMID 1343775。 ^ ユスポフ MM、ユスポワ GZ、バウコム A、リーバーマン K、アーネスト TN、ケイト JH、ノーラー HF 。「解像度 5.5 Å でのリボソームの結晶構造」。科学。292 (5518): 883–96。Bibcode : 2001Sci…292..883Y。土井:10.1126/science.1060089。PMID 11283358。S2CID 39505192。 ^ “リボソーム RNA | 遺伝学” . ブリタニカ百科事典。。
^ ゼモーラ G、ヴァルドシック C 。「生細胞におけるRNAの折り畳み」。RNA 生物学。7 (6): 634–41。土井:10.4161/rna.7.6.13554。PMC 3073324。PMID 21045541。 ^ フェルナンデス=トルネロ・C、モレノ=モルシージョ・M、ラシッド・UJ、テイラー・NM、ルイス・FM、グルーネ・T、他。。「14サブユニットRNAポリメラーゼIの結晶構造」。自然。502 (7473): 644–9。Bibcode : 2013Natur.502..644F。土井:10.1038/nature12636。PMID 24153184。S2CID 205235881。 ^ エンゲル C、セインズベリー S、チョン AC、コストレワ D、クレイマー P 。「RNAポリメラーゼIの構造と転写制御」。自然。502 (7473): 650–5。Bibcode : 2013Natur.502..650E。土井:10.1038/nature12712。hdl : 11858/00-001M-0000-0015-3B48-5。PMID 24153182。S2CID 205236187。 ^ Dutca LM、Gallagher JE、Baserga SJ 。「プレ 18S rRNA フォールディングに必要な最初の U3 snoRNA:プレ rRNA 塩基対相互作用が in vivo 化学プロービングによって明らかになりました。」核酸研究。39 (12): 5164–80。土井:10.1093/nar/gkr044。PMC 3130255。PMID 21349877。 ^ ウッドソン SA 。「RNAフォールディング経路とリボソームの自己集合」。化学研究のアカウント。44 (12): 1312–9。土井:10.1021/ar2000474。PMC 4361232。PMID 21714483。 ^ スローン KE、ワルダ AS、シャルマ S、エンティアン KD、ラフォンテーヌ DL、ボーンサック MT 。「リボソームの調整: 真核生物のリボソーム生合成と機能に対する rRNA 修飾の影響」。RNA 生物学。14 (9): 1138–1152。土井: 10.1080/15476286.2016.1259781。PMC 5699541。PMID 27911188。 ^ ジゴバ A、ドゥギンプディ S、ポレックス T、シェーファー M、コシュ M 。「25S rRNA のドメイン IV におけるメチル化のクラスターは、リボソームの安定性に必要です。 」RNA。20 (10): 1632–44。土井:10.1261/rna.043398.113。PMC 4174444。PMID 25125595。 ^ メトディエフ MD、レスコ N、パーク CB、カマラ Y、シー Y、ウィボム R、他。。「12S rRNA のメチル化は、哺乳動物のミトコンドリア リボソームの小サブユニットの生体内での安定性に必要です。」細胞の代謝。9 (4): 386–97。土井: 10.1016/j.cmet.2009.03.001。PMID 19356719。 ^ トンプソン M、ハウスラー RA、グッド PD、エンゲルケ DR 。「分散した tRNA 遺伝子の核小体クラスター化」。科学。302 (5649): 1399–401。Bibcode : 2003Sci…302.1399T。土井:10.1126/science.1089814。PMC 3783965。PMID 14631041。 ^ “rRNA の合成とプロセシング” .
^ スミット S、ウィドマン J、ナイト R (2007)。「進化速度はrRNAの構造要素によって異なります。 」核酸研究。35 (10): 3339–54。土井:10.1093/nar/gkm101。PMC 1904297。PMID 17468501。 ^ Chan JC、Hannan KM、Riddel K、Ng PY、Peck A、Lee RS、他。。「AKTはrRNA合成を促進し、c-MYCと協力してがんにおけるリボソーム生合成を刺激します。」サイエンスシグナリング。4 (188): ra56。土井:10.1126/scisignal.2001754。PMID 21878679。S2CID 20979505。 ^ Li S、Ibaragi S、Hu GF 。「前立腺がん治療の分子標的としてのアンジオゲニン」。現在のがん治療のレビュー。7 (2):83-90。土井:10.2174/1573394711107020083。PMC 3131147。PMID 21743803。 ^ Hoppe S、Bierhoff H、Cado I、Weber A、Tiebe M、Grummt I、Voit R 。「AMP 活性化プロテインキナーゼは rRNA 合成を細胞のエネルギー供給に適応させます。 」アメリカ合衆国国立科学アカデミーの議事録。106 (42): 17781–6。Bibcode : 2009PNAS..10617781H。土井:10.1073/pnas.0909873106。PMC 2764937。PMID 19815529。 ^ Liang XH、Liu Q、Fournier MJ 。「リボソームの解読中心におけるrRNA修飾の喪失は翻訳を損ない、pre-rRNAプロセシングを大幅に遅延させます。 」RNA。15 (9): 1716–28。土井:10.1261/rna.1724409。PMC 2743053。PMID 19628622。 ^ Larson K、Yan SJ、鶴見A、Liu J、Zhou J、Gaur K、他。。「ヘテロクロマチンの形成は長寿を促進し、リボソーム RNA 合成を抑制します。 」PLOS ジェネティクス。8 (1): e1002473。土井:10.1371/journal.pgen.1002473。PMC 3266895。PMID 22291607。
^ ガール T、バートレット MS、ロス W、ターンボー CL、ゴース RL (1997 年 12月)。「NTP濃度の開始による転写制御:細菌におけるrRNA合成」。科学。278 (5346): 2092–7。Bibcode : 1997Sci…278.2092G。土井:10.1126/science.278.5346.2092。PMID 9405339。 ^ 前田M、島田T、石浜A (2015-12-30). 「大腸菌における7つのrRNAプロモーターの強度と制御」。プロスワン。10 (12): e0144697。ビブコード: 2015PLoSO..1044697M。土井:10.1371/journal.pone.0144697。PMC 4696680。PMID 26717514。 ^ Gaal T、Bratton BP、Sanchez-Vazquez P、Sliwicki A、Sliwicki K、Vegel A、他。。「大腸菌の空間における離れた染色体座位の共局在:細菌の核小体」。遺伝子と発達。30 (20): 2272–2285。土井:10.1101/gad.290312.116。PMC 5110994。PMID 27898392。 ^ ウルフ、スティーブン (1993). 分子および細胞生物学。ISBN 978-0534124083。
^ Piir K、Paier A、Liiv A、Tenson T、Maiväli U 。「増殖中の細菌におけるリボソームの分解」。EMBOレポート。12 (5): 458–62。土井:10.1038/embor.2011.47。PMC 3090016。PMID 21460796。 ^ Brandman O、Hegde RS 。「リボソーム関連タンパク質の品質管理」。自然の構造および分子生物学。23 (1): 7-15。土井: 10.1038/nsmb.3147。PMC 4853245。PMID 26733220。 ^ 藤井一樹、北畠雅人、坂田哲也、宮田亜人、大野正樹 。「機能しないrRNAの除去におけるユビキチンの役割」。遺伝子と発達。23 (8): 963–74。土井: 10.1101/gad.1775609。PMC 2675866。PMID 19390089。 ^ ドノバン、ブリジット M. ジャレル、ケリー L. ラリヴィエール、フレデリック J. (2011-04-01)。「出芽酵母におけるストレス応答としての非機能的rRNA崩壊の研究」。FASEBジャーナル。25 (1_補足): 521.3。doi : 10.1096/fasebj.25.1_supplement.521.3 (2023 年 8 月 1 日に非アクティブ)。ISSN 0892-6638。 CS1 メンテナンス: DOI は 2023 年 8 月現在非アクティブです (リンク) ^ ラリヴィエール FJ、コール SE、フェルッロ DJ、ムーア MJ 。「成熟真核生物 rRNA の後期作用型品質管理プロセス」。分子細胞。24 (4): 619–26。土井:10.1016/j.molcel.2006.10.008。PMID 17188037。 ^ Michel JJ、McCarville JF、Xiong Y 。「適切な後期進行におけるSaccharomyces cerevisiae Cul8ユビキチンリガーゼの役割」。生物化学ジャーナル。278 (25): 22828–37。土井:10.1074/jbc.M210358200。PMID 12676951。S2CID 33099674。 ^ パイアー A、レピック M、スーサール A、テンソン T、マイヴァリ Ü 。「大腸菌におけるリボソーム分解に対するリボソーム活性部位およびサブユニット間接触における破壊の影響」。科学的報告書。5 : 7712。Bibcode : 2015NatSR…5E7712P。土井:10.1038/srep07712。PMC 4289901。PMID 25578614。 ^ 井出S、宮崎T、牧H、小林T 。「豊富なリボソーム RNA 遺伝子コピーがゲノムの完全性を維持する」。科学。327 (5966): 693–6。Bibcode : 2010Sci…327..693I。土井:10.1126/science.1179044。PMID 20133573。S2CID 206522454。
^ Quast C、Pruesse E、Yilmaz P、Gerken J、Schweer T、Yarza P、他。。「SILVA リボソーム RNA 遺伝子データベース プロジェクト: データ処理と Web ベースのツールの改良」。核酸研究。41 (データベースの問題): D590-6。土井:10.1093/nar/gks1219。PMC 3531112。PMID 23193283。 ^ Pruesse E、Peplies J、Glöckner FO 。「SINA: リボソーム RNA 遺伝子の正確なハイスループット複数配列アラインメント」。バイオインフォマティクス。28 (14): 1823 ~ 9 年。土井:10.1093/bioinformatics/bts252。PMC 3389763。PMID 22556368。
^ Wieland M、Berschneider B、Erlacher MD、Hartig JS 。「16S リボソーム RNA のアプタザイム媒介制御」。化学と生物学。17 (3): 236-42。土井:10.1016/j.chembiol.2010.02.012。PMID 20338515。 ^ ボーデン JR、ジョーンズ SW、インドゥルティ D、チェン Y、パプートサキス ET 。「非コード RNA とリボソーム RNA プロセシングに関与するクロストリジウム アセトブチリカムにおける新規の、おそらく合成の酸耐性機構のゲノム ライブラリーに基づく発見。」代謝工学。12 (3): 268–81。土井:10.1016/j.ymben.2009.12.004。PMC 2857598。PMID 20060060。 ^ トラウナー A、ロイド KE、ベネット MH、ヒングレーウィルソン SM、ウィリアムズ HD 。「休眠制御因子 DosR は、低酸素性マイコバクテリアのリボソームの安定性を制御します。 」生物化学ジャーナル。287 (28): 24053–63。土井:10.1074/jbc.m112.364851。PMC 3390679。PMID 22544737。 ^ マイヤー A、トッド C、ミケルセン NT、リーブ B 。「ソレノガストル(軟体動物)由来の急速に進化する 18S rRNA 配列は、標準的な PCR 増幅に抵抗し、軟体動物の置換率の不均一性について新たな洞察を与えます。」BMC進化生物学。10 (1): 70.土井: 10.1186/1471-2148-10-70。PMC 2841657。PMID 20214780。 ^ コール JR、チャイ B、マーシュ TL、ファリス RJ、ワン Q、クラム SA、他。。「リボソーム データベース プロジェクト (RDP-II): 定期的な更新と新しい原核生物の分類を可能にする新しいオートアライナーのプレビュー」。核酸研究。31 (1): 442–3。土井:10.1093/nar/gkg039。PMC 165486。PMID 12520046。 ^ Pruesse E、Quast C、Knittel K、Fuchs BM、Ludwig W、Peplies J、Glöckner FO (2007)。「SILVA: ARB と互換性のある品質チェックおよびアラインメントされたリボソーム RNA 配列データの包括的なオンライン リソース」。核酸研究。35 (21): 7188–96。土井:10.1093/nar/gkm864。PMC 2175337。PMID 17947321。 ^ ウェイド、M. Zhang, Y. (2005)、「結核菌における薬剤耐性のメカニズム」、結核と結核菌、米国微生物学会、115–140 ページ、doi : 10.1128/9781555817657.ch8、ISBN 9781555817657、S2CID 36002898 ^ Long KS、Poehlsgaard J、Hansen LH、Hobbie SN、Böttger EC、Vester B 。「リボソームペプチジルトランスフェラーゼ中心における単一の23S rRNA変異は、薬物結合ポケットの混乱により、スメグマ菌にバルネムリンおよび他の抗生物質に対する耐性を与える。」分子微生物学。71 (5): 1218–27。土井: 10.1111/j.1365-2958.2009.06596.x。PMID 19154331。S2CID 23728518。 ^ チュソンD (2013). 「プレリボソーム RNA に由来する非典型的な機械感受性 microRNA-712 は、内皮炎症とアテローム性動脈硬化を誘発します。 」ネイチャーコミュニケーションズ。4 : 3000。Bibcode : 2013NatCo…4.3000S。土井: 10.1038/ncomms4000。PMC 3923891。PMID 24346612。
外部リンク
16S rRNA、BioMineWiki
リボソームデータベースプロジェクトⅡ
米国国立医学図書館の医療主題見出し(MeSH)のリボソーム + RNA
SILVA rRNA データベース プロジェクト(真核生物 (18S) および LSU (23S/28S) も含む)
ビデオ: rRNA: 配列、機能、合成
Halococcus morrhuae (古細菌) 5S rRNA · “